NGS 데이터로 SNP와 Indel 분석하기
- Posted at 2012/04/20 14:04
- Filed under 제품소식
Variation study
'시퀀싱 비용의 절감’ 이라는 장점을 갖는 NGS 기술과 함께 이슈가 되고 있는 분야가 ‘개인 맞춤형 진단’입니다. 질병, 체질 등 모든 표현형의 근간이 되는 DNA의 서열 정보를 알아내어 비교함으로써 개인 간의 차이와 질병의 원인을 알아낼 수 있는 시도를 진행할 수 있게 된 것입니다. NGS 데이터를 이용해 수행할 수 있는 variation 분석으로 SNP, small insertion/deletion polymorphisms, structure variation 분석이 있습니다.
분석 방법은 대략 비슷한데,
1. Reference 서열에 mapping
2. Variation(SNP, Indel, etc)찾기
3. Public DB 데이터와 비교
와 같은 순서로 볼 수 있습니다.
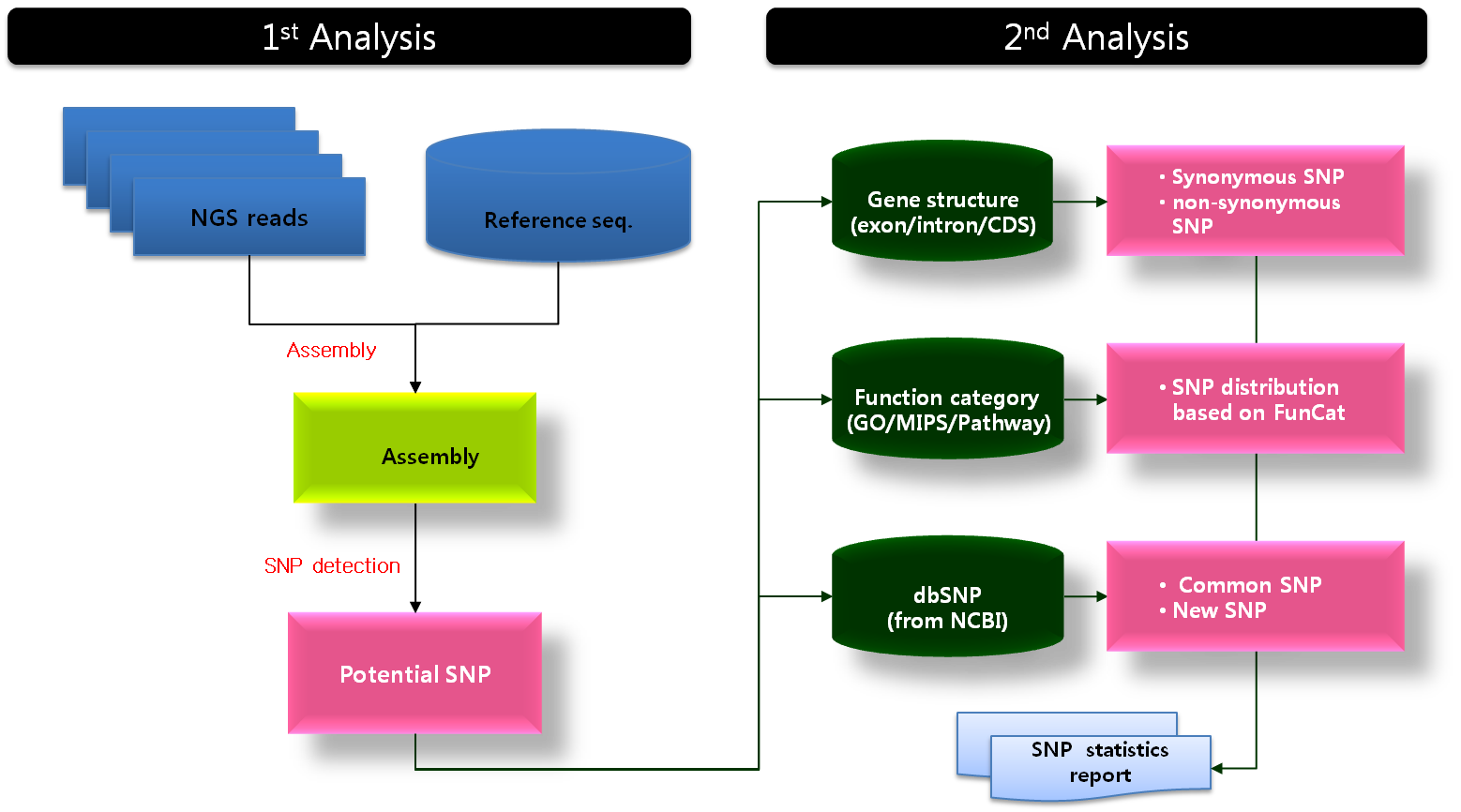 보통 mapping을 하기 전에 read 들을 quality나 시퀀싱 기기에 잠재적인 오류를 줄이기 위한 filtering을 먼저 진행하지만 여기선 언급하지 않겠습니다. Reference
서열과 read가 준비되면 reference assembly를 합니다. 그리고 그 결과로 나온 mapping 데이터에서
reference 서열과 consensus 서열, 그리고 consensus 서열을 만들어낸 read들의 서열 데이터를 모두
종합해서 SNP나 Indel을 찾아냅니다.
보통 mapping을 하기 전에 read 들을 quality나 시퀀싱 기기에 잠재적인 오류를 줄이기 위한 filtering을 먼저 진행하지만 여기선 언급하지 않겠습니다. Reference
서열과 read가 준비되면 reference assembly를 합니다. 그리고 그 결과로 나온 mapping 데이터에서
reference 서열과 consensus 서열, 그리고 consensus 서열을 만들어낸 read들의 서열 데이터를 모두
종합해서 SNP나 Indel을 찾아냅니다.
SNP 찾기
먼저 SNP를 찾는 방법에 대해 정리해 보고자 합니다. 사실 SNP를 찾는 소프트웨어들이 하는 일은 reference 서열과 consensus 서열이 서로 다른 position을 찾은 후, 그 position의 consensus 서열을 구성한 read들의 quality나 coverage, 그리고 구성 비율 등의 기준을 정하고 그 기준을 통과하는 position을 찾아 정리해주는 역할을 합니다.
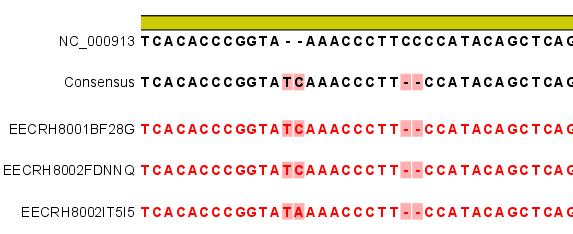
아래 그림에서 노란색으로 강조된 세로 열을 보면 consensus 서열(black)이 ‘A’ 이지만 이 서열에 해당하는 read들을 보면 일부 ‘C' 가 보입니다. Window size라는 것은 SNP를 찾는데 특정 position에 해당 하는 read의 서열 주변 영역을 말합니다. 예를 들어서 window size가 11이라고 정해지면 특정 position의 양옆으로 5bp 씩 확장한 11bp를 의미합니다. 만약 read의 해당 position이 끝 부분이어서 한쪽으로 확장할 영역이 5bp 미만일 경우 부족한 만큼 반대쪽 영역으로 확장하여 비대칭한 형태로 window size가 설정 됩니다. 이 window size을 대상으로 quality나 gap, mismatch 개수를 계산한 다음 해당 position의 consensus 서열을 결정하게 만든 read들의 정보가 믿을 수 있는지에 대한 filtering을 하게 됩니다. 만약 해당 position을 서열을 결정하는 read 서열 주변(window size) 영역의 quality가 낮거나 gap 또는 mismatch가 많다면, 그 read의 서열은 신뢰하기 어렵기 때문에 SNP를 결정 할 때 제외해야 할 것입니다.
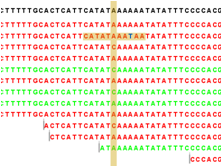 이렇게 믿을 수 있는 read 정보만 남겨놓은 다음에는 read 서열의 frequency를 계산하여, 해당 position에
대하여 reference 서열과 다른 read의 서열의 frequency에 대한 기준을 정해 SNP를 찾아냅니다. 예
를 들어 위 그림의 강조된 부분에 mapping 된 14개 read 중 8개의 read는 'A'이지만 4개의 read는 C를
가리키고 있습니다. A와 C의 frequency는 66.67%와 33.33%입니다. 만약 reference 서열이 'A'이고 기준
frequency를 30% 라고 정했다면, 이 position은 SNP로 찾아질 것입니다. 이 frequency에 대한 기준은
sample을 어떻게 준비했느냐에 따라 달라집니다. 예를 들어 이배체 종의 sample을 그대로 시퀀싱 했다면 부모로 받은 서로
다른 두 개의 형질이 섞인채로 시퀀싱 되어 실제 SNP를 찾기 힘들기 때문입니다.
이렇게 믿을 수 있는 read 정보만 남겨놓은 다음에는 read 서열의 frequency를 계산하여, 해당 position에
대하여 reference 서열과 다른 read의 서열의 frequency에 대한 기준을 정해 SNP를 찾아냅니다. 예
를 들어 위 그림의 강조된 부분에 mapping 된 14개 read 중 8개의 read는 'A'이지만 4개의 read는 C를
가리키고 있습니다. A와 C의 frequency는 66.67%와 33.33%입니다. 만약 reference 서열이 'A'이고 기준
frequency를 30% 라고 정했다면, 이 position은 SNP로 찾아질 것입니다. 이 frequency에 대한 기준은
sample을 어떻게 준비했느냐에 따라 달라집니다. 예를 들어 이배체 종의 sample을 그대로 시퀀싱 했다면 부모로 받은 서로
다른 두 개의 형질이 섞인채로 시퀀싱 되어 실제 SNP를 찾기 힘들기 때문입니다.
이렇게 염기서열 상에서의 SNP를 찾고난 다음에는 이 서열이 발현 단계에서 아미노산 서열의 변화까지 일으키는 non-synonymous SNP인지 확인해야 합니다.
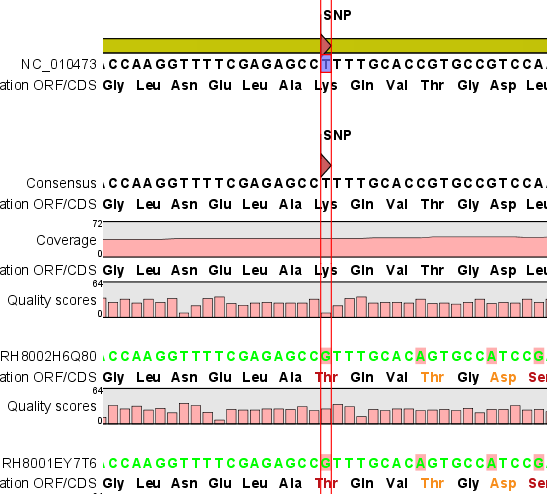
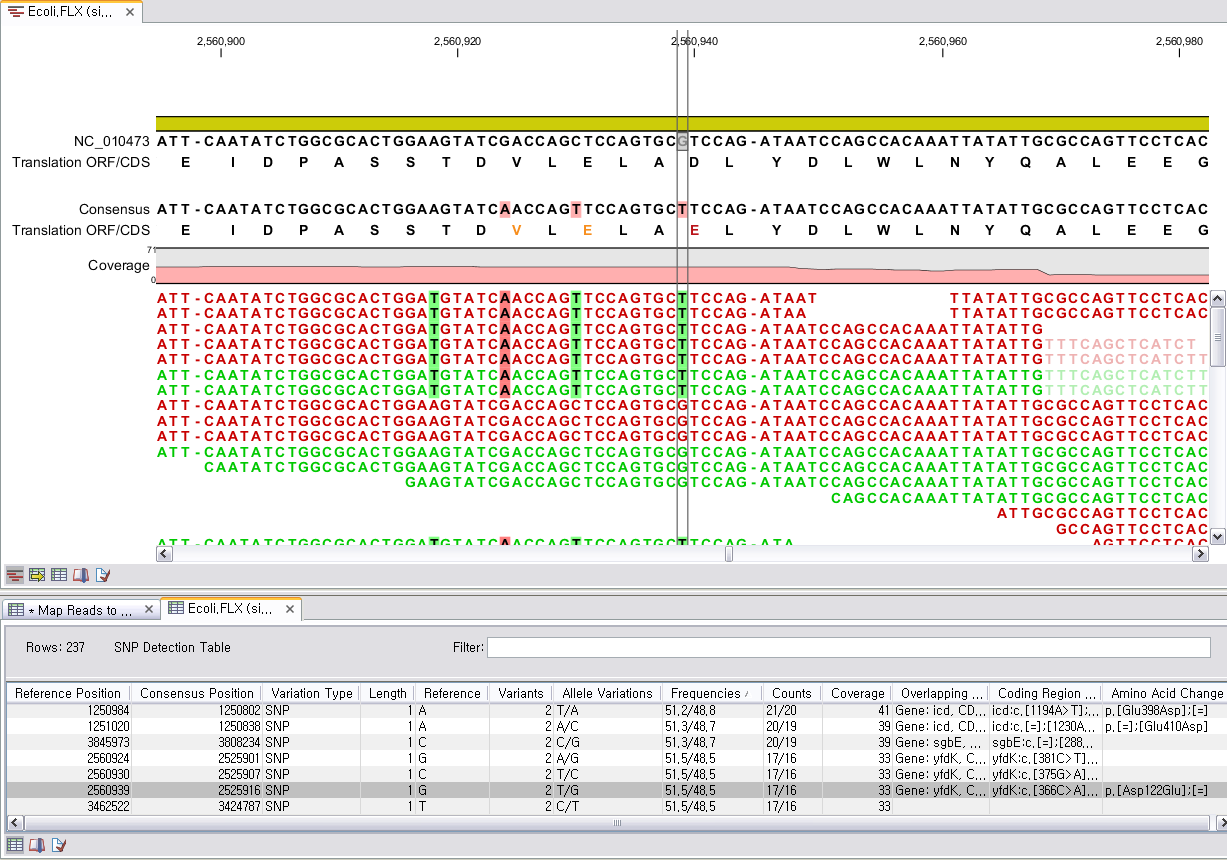
DIP 찾기
편의상 Deletion/Insertion(gap) polymorphism을 줄여서 DIP라고 부르겠습니다. DIP를 찾는 것도 SNP를 찾는 방법과 유사합니다. Reference 서열과 비교해서 consensus 서열에 나타난 insertion이나 deletion이 나타난 자리를 찾는 것 입니다. 이 때 SNP와 마찬가지로 DIP가 나타난 consensus 서열의 근거가 되는 read의 수나 frequency를 기준으로 DIP를 선별해 낼 수 있습니다. DIP의 경우 1~2bp로 인해 해당 유전자의 ORF 전체가 바뀌게 되므로 관련된 유전자와 관련된 구조적 변화나 질병 등에 대한 연구가 함께 필요합니다.
* 아래 이메일 주소로 연락 주시면 CLC Genomics Workbench의 모든 기능을 사용할 수 있는 데모 라이센스를 제공해 드리오니 많은 이용 바랍니다.
- codes@insilicogen.com
'시퀀싱 비용의 절감’ 이라는 장점을 갖는 NGS 기술과 함께 이슈가 되고 있는 분야가 ‘개인 맞춤형 진단’입니다. 질병, 체질 등 모든 표현형의 근간이 되는 DNA의 서열 정보를 알아내어 비교함으로써 개인 간의 차이와 질병의 원인을 알아낼 수 있는 시도를 진행할 수 있게 된 것입니다. NGS 데이터를 이용해 수행할 수 있는 variation 분석으로 SNP, small insertion/deletion polymorphisms, structure variation 분석이 있습니다.
분석 방법은 대략 비슷한데,
1. Reference 서열에 mapping
2. Variation(SNP, Indel, etc)찾기
3. Public DB 데이터와 비교
와 같은 순서로 볼 수 있습니다.

SNP 찾기
먼저 SNP를 찾는 방법에 대해 정리해 보고자 합니다. 사실 SNP를 찾는 소프트웨어들이 하는 일은 reference 서열과 consensus 서열이 서로 다른 position을 찾은 후, 그 position의 consensus 서열을 구성한 read들의 quality나 coverage, 그리고 구성 비율 등의 기준을 정하고 그 기준을 통과하는 position을 찾아 정리해주는 역할을 합니다.
아래 그림에서 노란색으로 강조된 세로 열을 보면 consensus 서열(black)이 ‘A’ 이지만 이 서열에 해당하는 read들을 보면 일부 ‘C' 가 보입니다. Window size라는 것은 SNP를 찾는데 특정 position에 해당 하는 read의 서열 주변 영역을 말합니다. 예를 들어서 window size가 11이라고 정해지면 특정 position의 양옆으로 5bp 씩 확장한 11bp를 의미합니다. 만약 read의 해당 position이 끝 부분이어서 한쪽으로 확장할 영역이 5bp 미만일 경우 부족한 만큼 반대쪽 영역으로 확장하여 비대칭한 형태로 window size가 설정 됩니다. 이 window size을 대상으로 quality나 gap, mismatch 개수를 계산한 다음 해당 position의 consensus 서열을 결정하게 만든 read들의 정보가 믿을 수 있는지에 대한 filtering을 하게 됩니다. 만약 해당 position을 서열을 결정하는 read 서열 주변(window size) 영역의 quality가 낮거나 gap 또는 mismatch가 많다면, 그 read의 서열은 신뢰하기 어렵기 때문에 SNP를 결정 할 때 제외해야 할 것입니다.

이렇게 염기서열 상에서의 SNP를 찾고난 다음에는 이 서열이 발현 단계에서 아미노산 서열의 변화까지 일으키는 non-synonymous SNP인지 확인해야 합니다.


DIP 찾기
편의상 Deletion/Insertion(gap) polymorphism을 줄여서 DIP라고 부르겠습니다. DIP를 찾는 것도 SNP를 찾는 방법과 유사합니다. Reference 서열과 비교해서 consensus 서열에 나타난 insertion이나 deletion이 나타난 자리를 찾는 것 입니다. 이 때 SNP와 마찬가지로 DIP가 나타난 consensus 서열의 근거가 되는 read의 수나 frequency를 기준으로 DIP를 선별해 낼 수 있습니다. DIP의 경우 1~2bp로 인해 해당 유전자의 ORF 전체가 바뀌게 되므로 관련된 유전자와 관련된 구조적 변화나 질병 등에 대한 연구가 함께 필요합니다.

* 아래 이메일 주소로 연락 주시면 CLC Genomics Workbench의 모든 기능을 사용할 수 있는 데모 라이센스를 제공해 드리오니 많은 이용 바랍니다.
- codes@insilicogen.com
Posted by 人Co
- Response
- No Trackback , No Comment
- RSS :
- https://post-blog.insilicogen.com/blog/rss/response/108
Trackback URL : 이 글에는 트랙백을 보낼 수 없습니다


