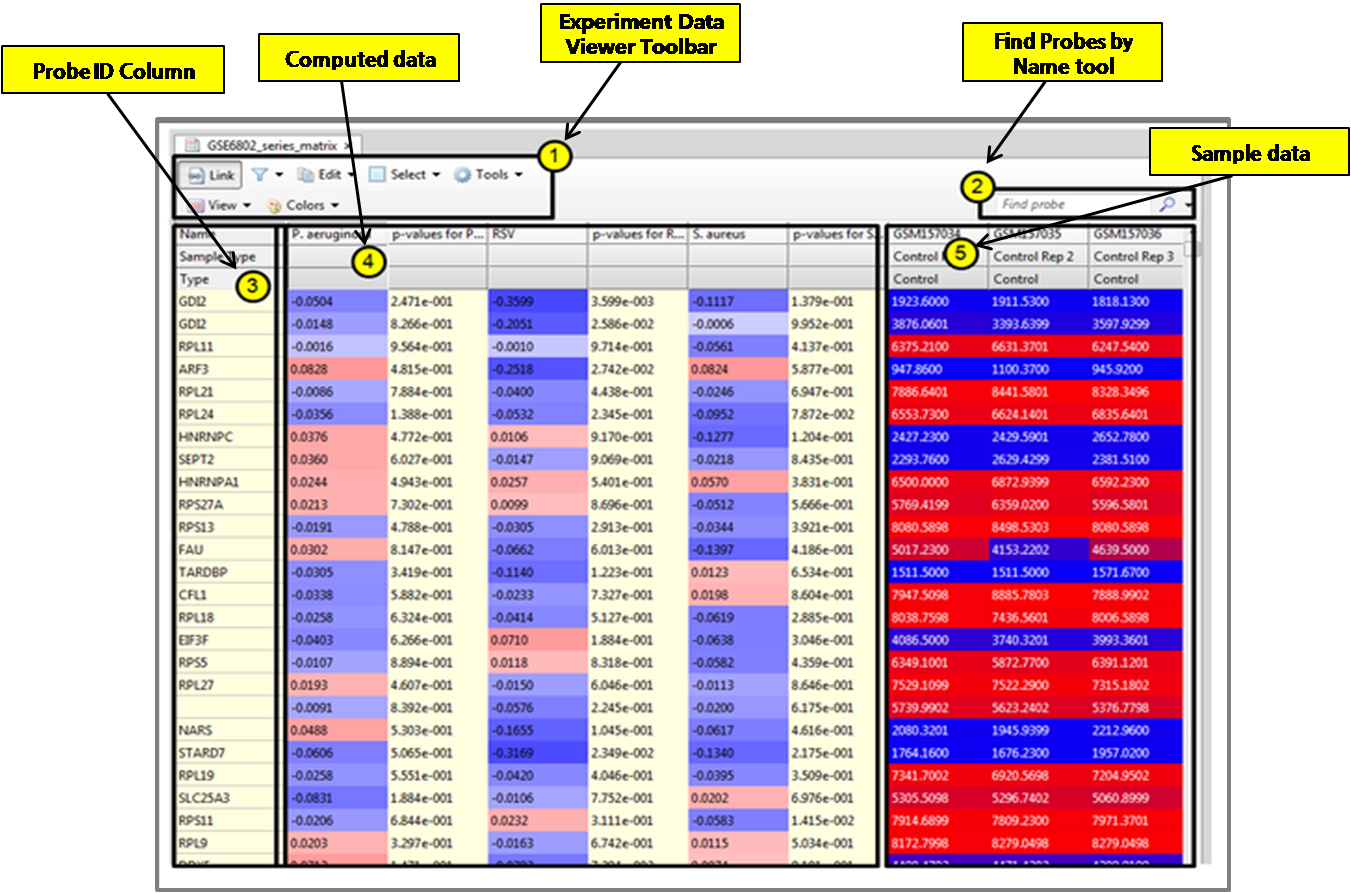
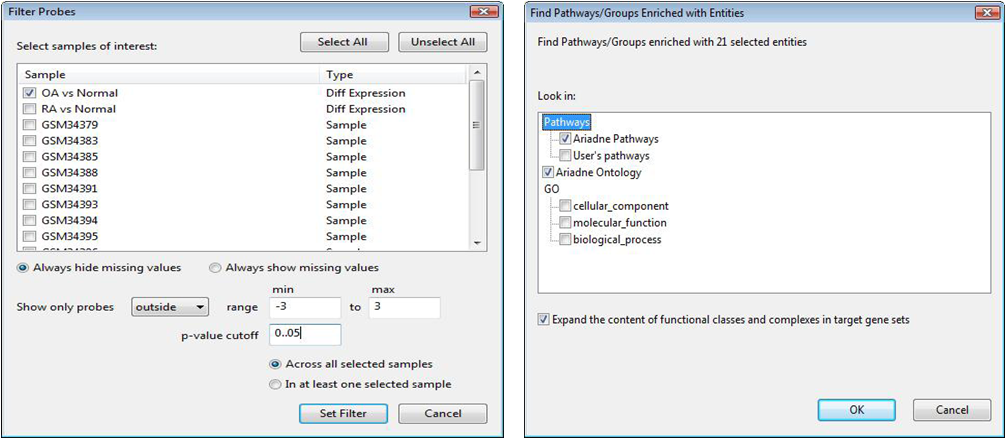
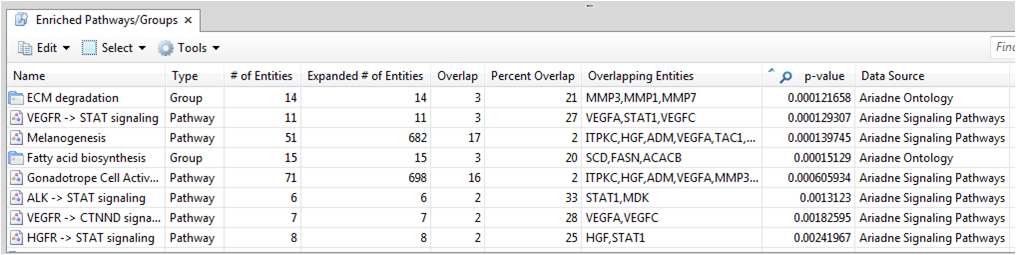
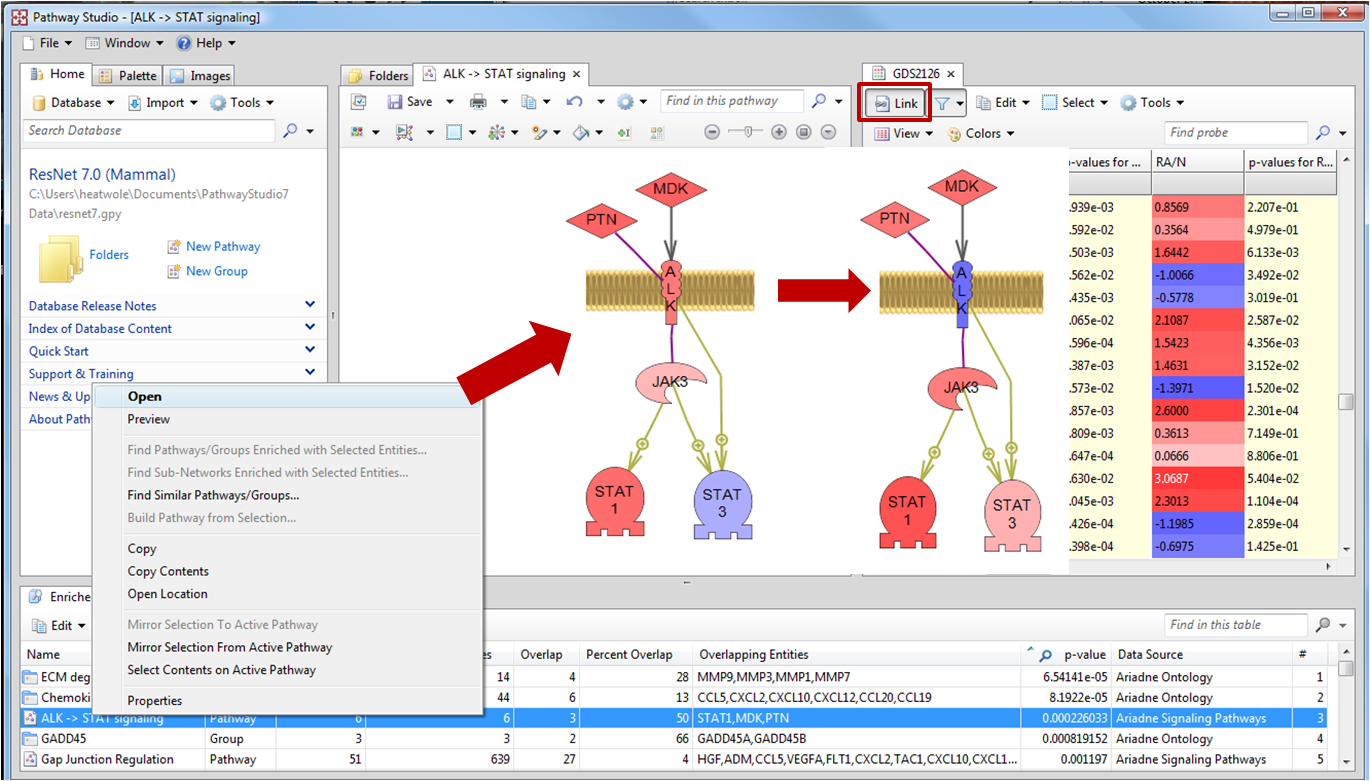
연재 순서
1. PathwayStudio 소개
2. 문헌정보를 활용한 유전자 네트워크 분석
3. Chip 실험 데이터에서의 유전자 네트워크 분석
4. Drug 발굴을 위한 지식 데이터베이스 ChemEffect
카레 성분의
커큐민(cucumin)이 전립선암을 억제한다고?
미국에는 해마다
전립선암 환자가 증가하고 있는데 반해 인도인들은 전립선암 환자가 거의 발생하지 않고 있다. 유전적인 요인도 있겠지만, 식습관의
차이가 전립선 암환자의 발생을 유도하거나 억제하지 않을까라는 단순한 궁금증을 가지고 카레와 전립선암과의 연관관계에 대한 연구를
시작하고자 한다. 이때 우리는 “인도인들이 즐겨먹는
카레의 주성분에서 전립선암을 억제하는 상호기작이 있을 것이다”라고 가정할 수 있다. 그럼 카레의 주성분은 무엇일까? 카레의 색깔이
노란색인 것은 커큐민이라는 성분때문인데, 이 커큐민이 카레의 주성분이다. 전립선암과 우리가 즐겨 먹는 카레의 주성분인 커큐민은
상호 어떤 관계가 있을까? 실험을 통해서 일일이 검증을 해야 확인할 수 있겠지만, PubMed와 같이 과학 문헌
데이터베이스에서 커큐민 성분에 영향을 미치는 유전자에 관련된 논문과 전립선암에 관여하는 유전자에 대한 논문을 찾은 후
‘커큐민-유전자-전립선암’과의 관계를 유추할 수 있다.
이와 같이 신약을
발굴하기 위해서 바이오마커를 찾거나, 특정 질병에 관여하는 유전자들이 무엇인지 알고자 할 경우, 또는 DNA Chip 분석을
통해서 얻어지는 차등 발현 유전자들이 공통적으로 관여하는 질병을 찾거나, 유전자들의 상호 연관관계를 알고자 할 경우에 사용되는
유용한 프로그램 가운데
PathwayStudio라
는 프로그램을 소개하고자 한다.
Pathway Studio
시스템 구성
Pathway Studio는 세 가지 시스템으로 구성되어 있다.
- MedScan: 자연어처리기술(NaturalLanguageProcessing)
을 이용한 과학 문헌의 전문화된 텍스트 마이닝프로그램, 단백질 중심의 생물학적 연관관계 추출
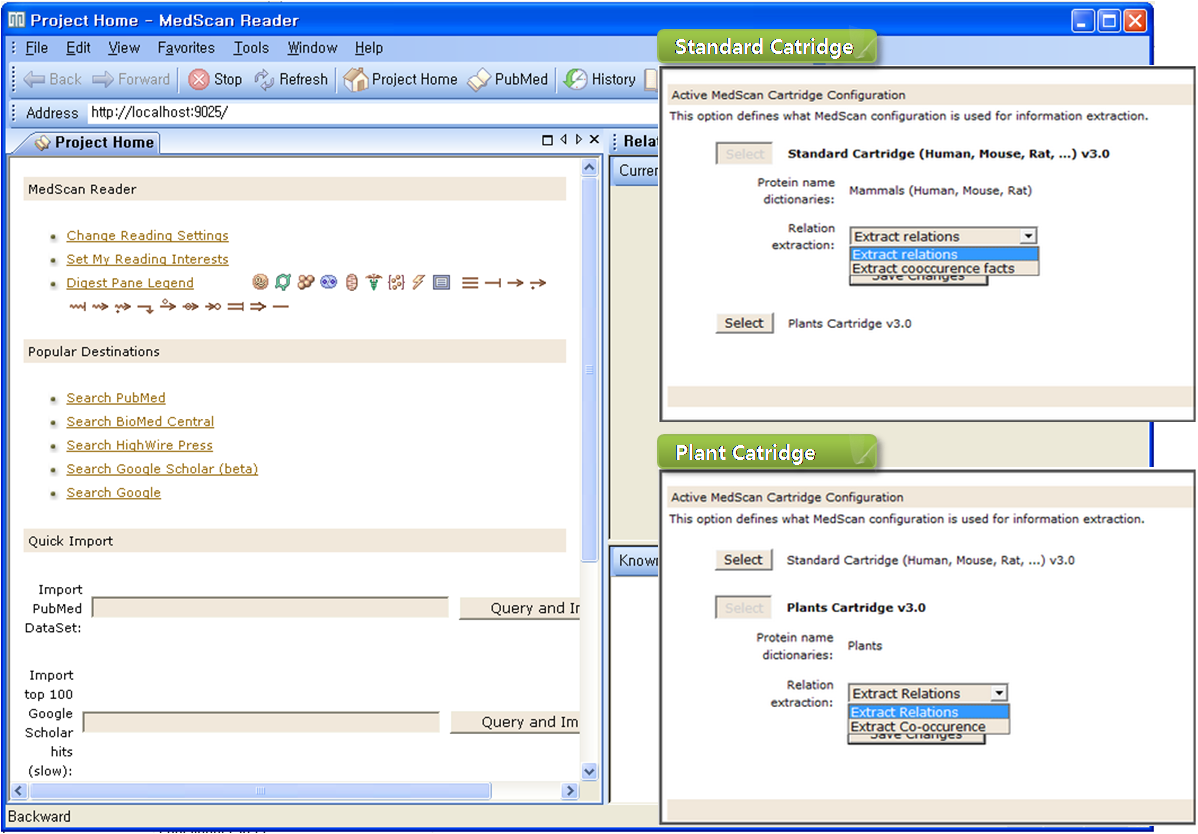
- ResNet Database: MedScan을 이용하여 PubMed와 Interaction 관련
저널에서 추출한 Mammalian, Plant의 생물학적 네트워크 정보를 생물학 전문가에 의해 재검증한 데이터베이스
- PathwayStudio:
MedScan과ResNet Database를 통해
추출된 데이터를 이용하여 Pathway를 편집 할 수 있도록 제공되는 사용자-친숙한 인터페이스
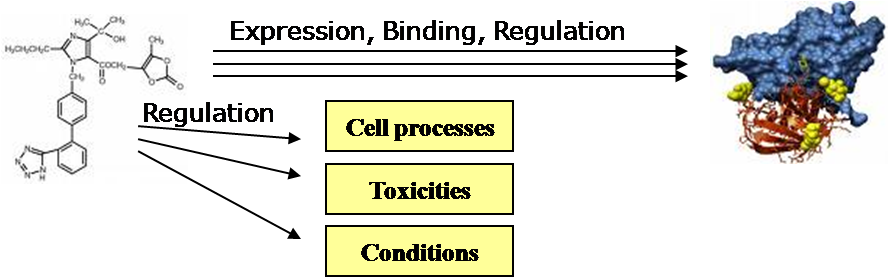
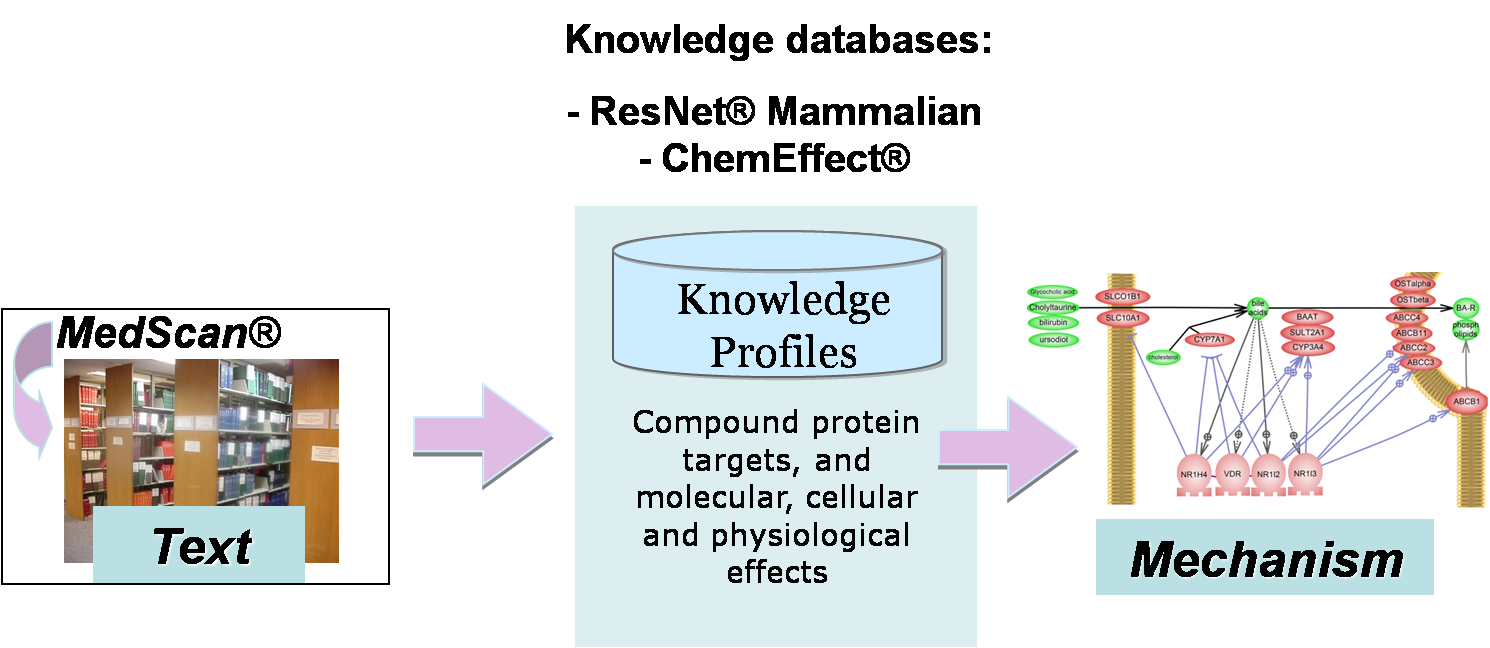
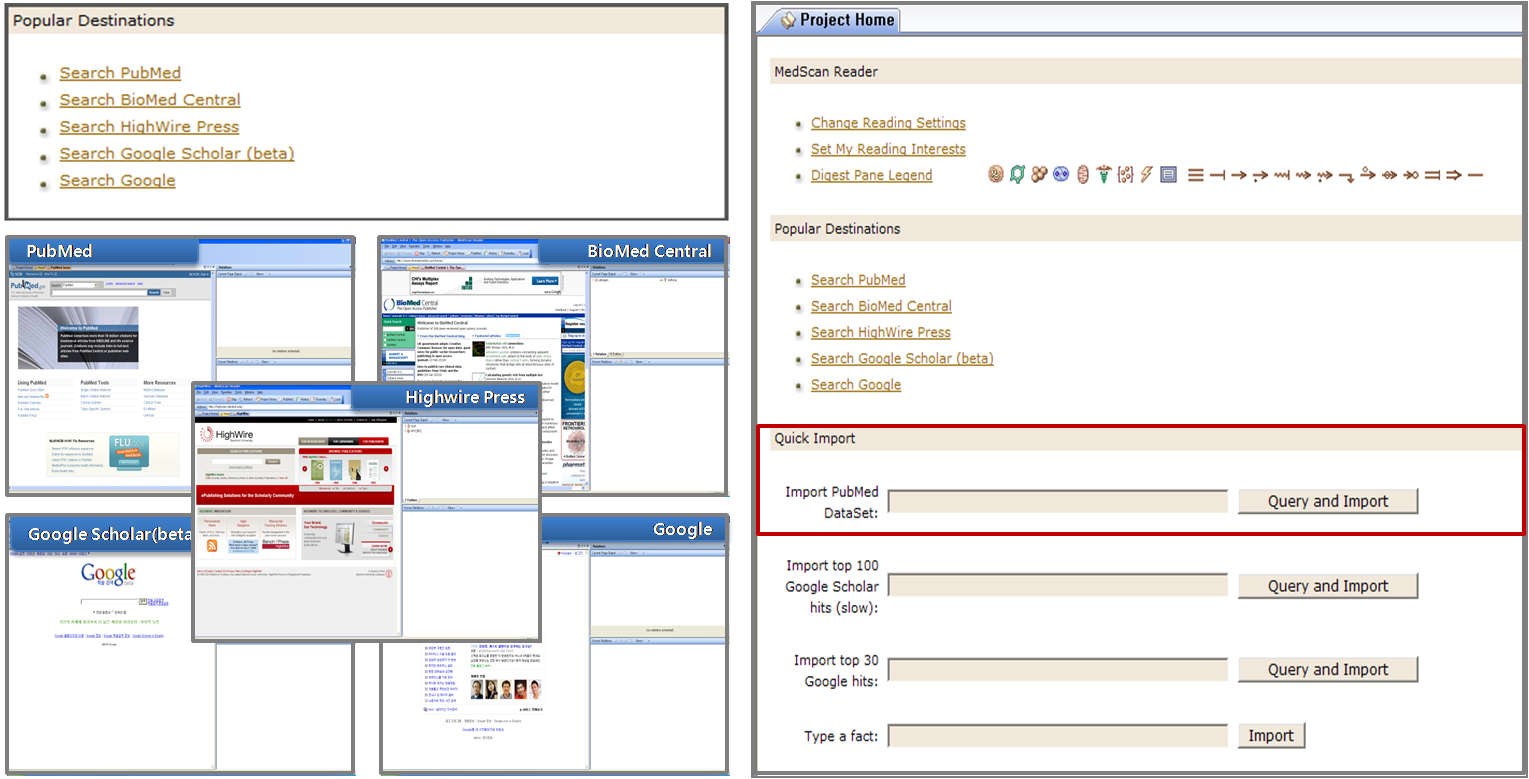
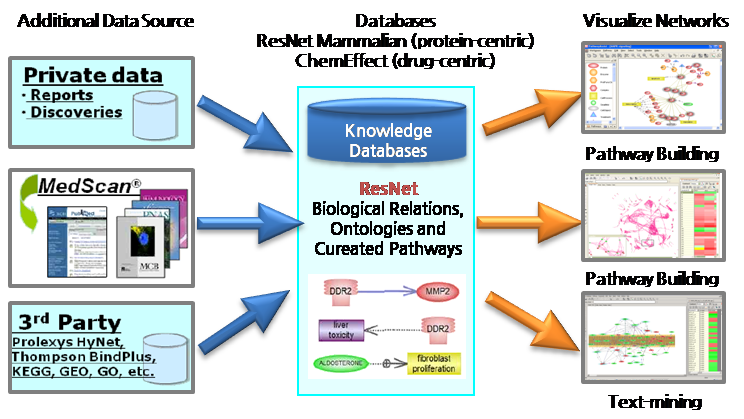
그림 1. 시스템의 구성. 1) 자연어 처리 기술을 응용하여 문헌에서 생물학적인 데이터를 추출하는 MedScan Reader 2)
추출된 데이터를 데이터베이스화 한 ResNetResNet
Database는 Mammalian과 Plant 두 가지로 구분되어진다. 3) 데이터베이스의 정보를 그래픽 형태로 pathway를
생성하고 편집할 수 있는 인터페이스를 제공하는 PathwayStudio
Database. Pathway Studio
응용분야
Pathway Studio는
- 유전자 발현 데이터 또는 high throughput 데이터를 해석할 때,
- pathway를 설계, 확장하고 분석 할 때,
- 유전자, 단백질, cell processes, 질병 사이의 관계를 찾을 때,
- publication-quality pathway 다이어그램을 그릴 때,
- 문헌 정보에서 바이오마커와 drug 후보군을 찾을 때,
와 같은 다양한 연구 분야에서 사용되고 있으며, 수많은 연구자들이 Pathway Studio를 이용하여 분석한 결과 및 방법들을
논문으로 투고하고 있다. ARIADNE사의 홈페이지((
http://www.ariadnegenomics.com/technology-research/publications/))
에는 아래와 같은 카테고리 별로 PathwayStudio를
이용하여 투고된 논문 정보를 바로 확인할 수 있다.
- Epigenetic studies
- Pathway Analysis
- Analysis of gene expression microarray data
- Anayisis of proteomics data
- Drug discovery
- Human genetics
- Toxicogenomics
- Biomarkers
- Neuroscience
- Text mining
- Model organisms
- Plants
Pathway Build 방법
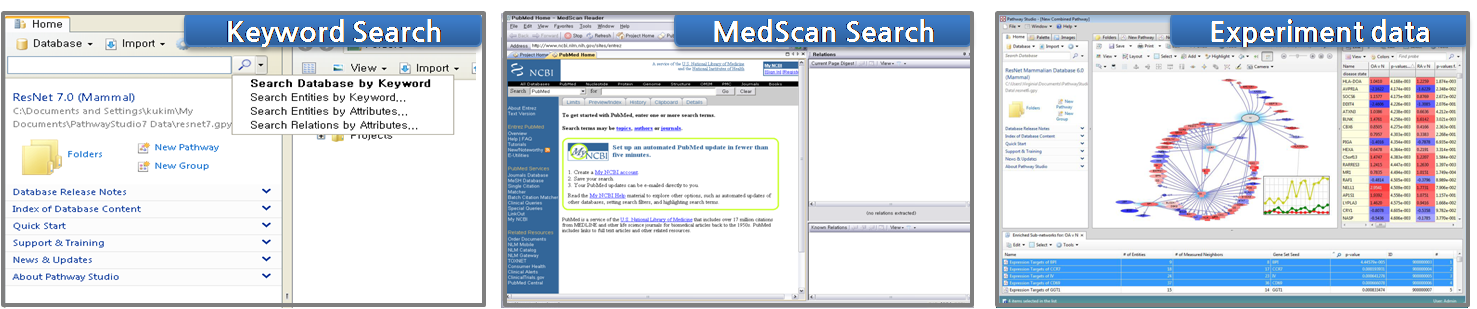
과연, 그렇다면 Pathway Studio에서는 어떤 방법으로 pathway를 그릴 수 있을까? pathway를 그릴 수 있는
방법은 아래 그림과 같이 크게 3가지 정도로 나누어 볼 수 있다. 첫 번째로 내가 알고 있는 유전자 하나 혹은 여러 개의 유전자
목록을 검색하여 엔티티들 사이에 어떤 관계가 있는지 pathway를 직접 그려가면서 확인하는
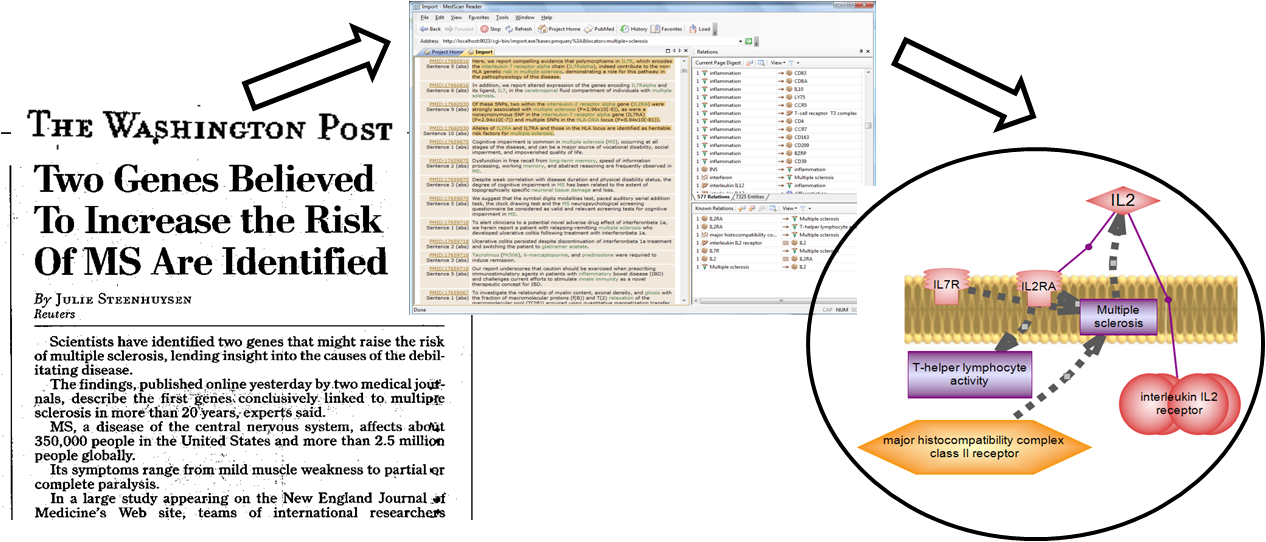
검색을 통한 방법이
다. 두 번째로는 어떠한 주제로 연구를 할 때 기존에 밝혀져 있는 문헌에 대해 리뷰하는 과정을 거치게 되는데 이 때 관련 문헌을
모두 검색하고 거기에서 보고자 하는 정보들을 추출 할 수 있다. 이렇게 추출된 정보들은
문헌을 통한 검색으
로 추출되었기 때문에 신뢰도가 높은 정보를 제공할 뿐만 아니라 Pathway Studio를 통해서 그들 간의 pathway도 그려
볼 수 있고, 그것을 더 확장해 나가면서 새로운 의미를 도출 할 수도 있다. 마지막으로
실제 실험을 통해 나온
데이터를 입력하고 통계 분석을 한 뒤
통계적으로 유의한 유전자들 사이의 관계를
pathway로 그려보고 발현양상을 살펴보는 방법이 있다.
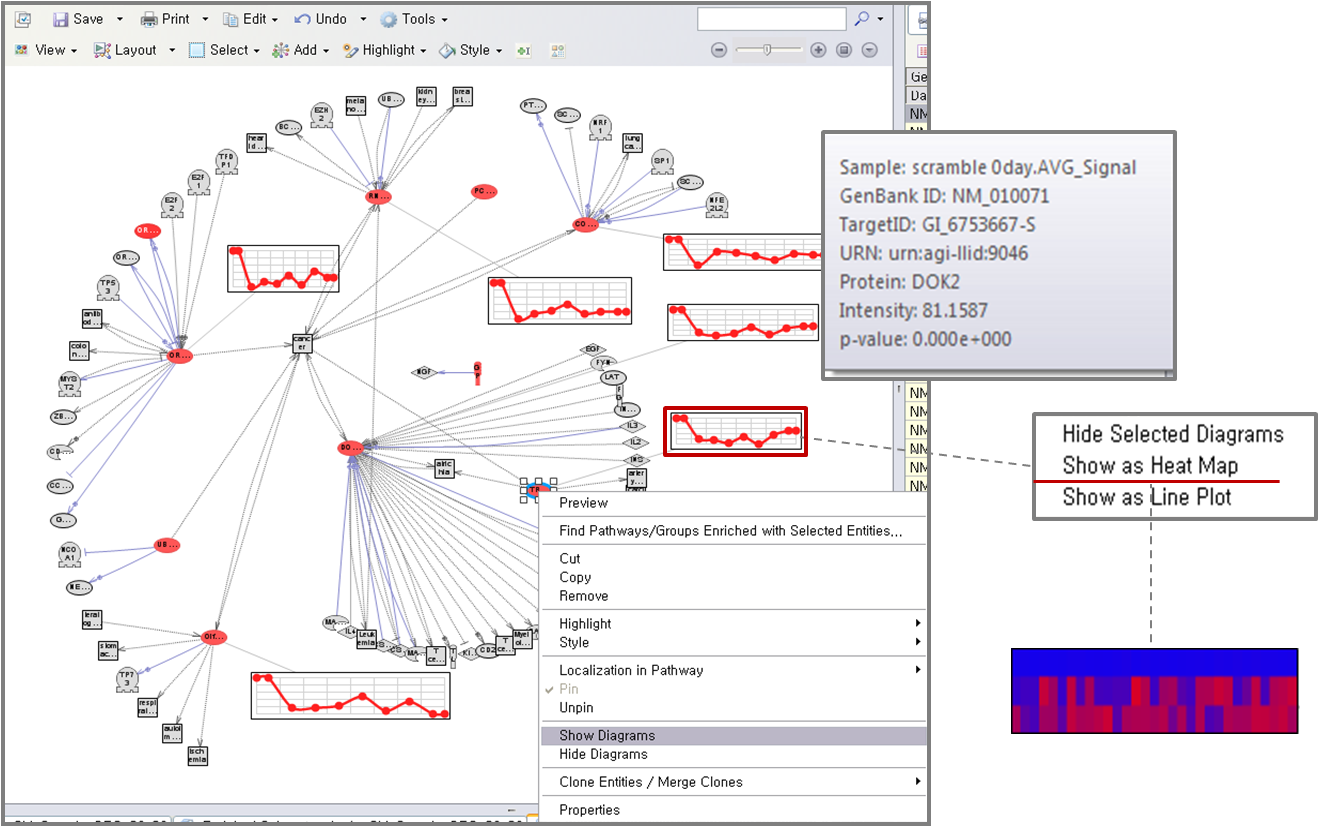
그림 2. pathway build 방법 1) 유전자 검색을 통한 방법 2) MedScan 문헌 검색을 통한 방법
3) Import한 실험 데이터를 이용하는 방법 Pathway 편집
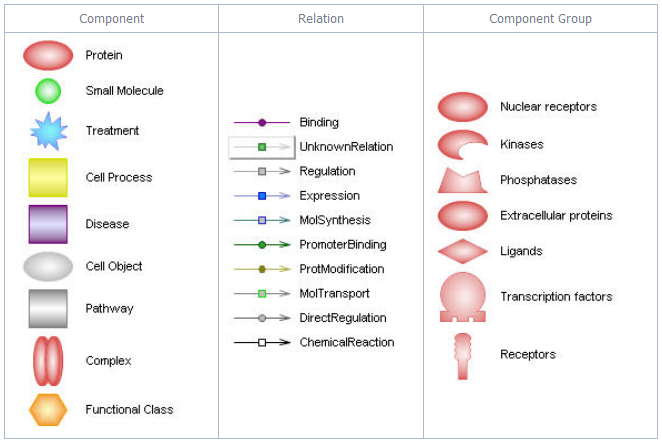
위의 세 가지 방법을 통해 pathway를 그릴 때에는 pathway를 보기 쉽게 편집하는 것 또한 중요하다. Pathway
Studio에서는 pathway를 그릴 때 편집하기 쉬운 인터페이스를 제공하고 있을 뿐 아니라 서로 다른 Entity와
Relation을 한 눈에 구별 할 수 있도록 그것을 다양한 모양과 색으로 표현하고 있다. 완성된 pathway를 이미지로 저장할
때에는 아래 그림과 같이 Entity와 Relation 정보를 범례로 포함하여 저장 할 수 있어 pathway를 처음 보는
사람이더라도 쉽게 그 관계를 이해 할 수 있다. 또한 그려진 pathway에서 Relation을 나타내는 화살표에 마우스를
가져가면 Entity들 간에 어떤 관계에 있는지 그리고 그 관계를 뒷받침 할 수 있는 관련 문장이 어떤 문헌에서 추출 되었는지와
같은 정보를 제공해 준다.
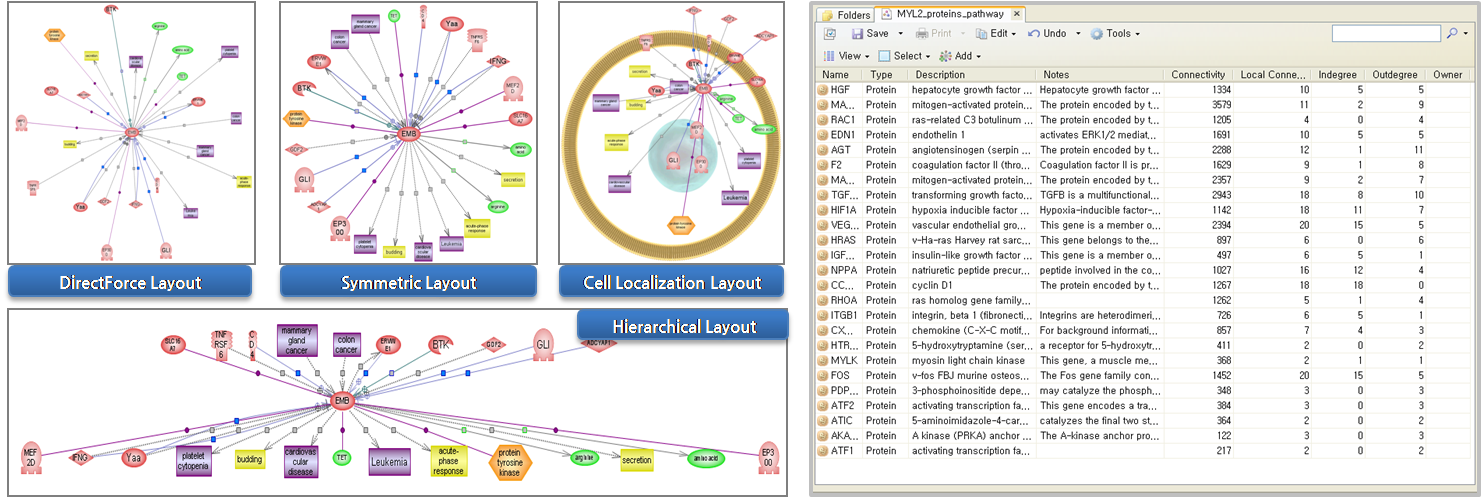
그림 3. Entity, Relation, component group의 종류Pathway layout
완성된 pathway는 layout을 바꾸어 가면서 볼 수 있다. Pathway Studio에서는 DirectForce
layout, Symmetric layout, Cell Localization layout, Hierachical layout과
같이 다양한 layout을 제공하고 있다. layout을 변경을 통해서 보다 새로운 의미를 찾을 수 있을 것이다.
- DirectForce
layout : Entity를 중심으로 관련 정보를 축 방향으로 나타내주는 layout
- Symmetric layout : Entity를 중심으로 관련 정보를 대칭 형태로 나타내주는 layout
- Cell Localization layout : pathway의 Entity들이 Cell 안에서 어떤 곳에 위치하는지 보여주는
layout
- Hierachical layout : Entity를 중심으로 유전자를 조절하는 up-regulation 또는 공통적으로 작용하는
down-regulation 정보를 계층적인 구조로 보여주는 layout
layout을 통해 그랙픽 형식으로 검토하는 방법 이외에도 Entity table, Relation table 보기 기능도
지원하고 있다. Table 보기에서는 Entity와 Relation 정보 이외에도 다양한 annotation 정보도 함께
제공하고, 테이블의 컬럼도 사용자의 편의에 맞게 선택적으로 customizing 하여 볼 수 있도록 되어 있다.
그림 4. pathway view 방식 1) pathway 그래프 보기의 다양한 layout 형태
2) Entity, Relation 테이블 형태의 보기 방식 Pathway data
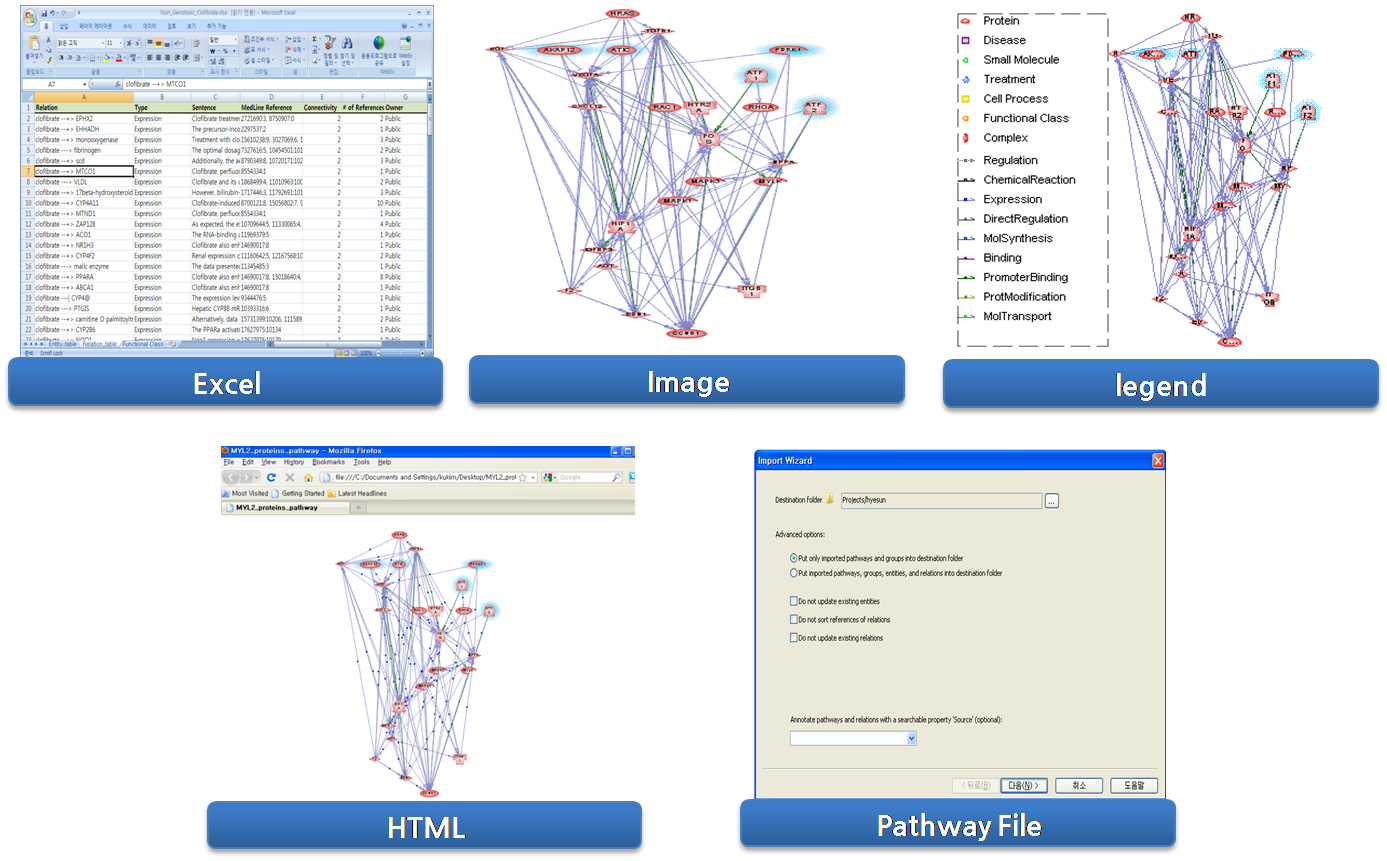
export
pathway는 다양한 형태로도 저장이 가능하다. 그래프 형태의 pathway는 .gif, .jpg, .png, .tif,
.bmp 5가지 확장자의 이미지 파일로 저장 할 수 있다. 이미지로 저장을 할 때에는 범례를 포함하여 저장 할 수 있는데
pathway를 문헌에 효율적으로 이용할 수 있도록 이미지의 넓이, 높이의 크기와 DPI 해상도까지 지정할 수 있다.
Entity와 Relation 정보의 table 보기는 엑셀의 형태로 저장 할 수 있어 차후에 2차 분석을 할 때 유용하게 사용할
수 있다. 이 밖에도 웹 문서인 HTML 형태로 저장을 하면 Pathway Studio가 설치되어 있지 않은 곳에서도 웹을 통해
데이터들을 모두 볼 수 있으며 Pathway Studio의 고유 파일 형식인 .gpp 파일 포맷으로 저장을 할 경우에는
Pathway Studio가 설치되어 있는 사용자 간의 혹은 pathway 데이터를 백업 할 때 사용할 수 있다.
그림 5. Pathway 데이터 export 방식.
2010년 5월 24일 현재 Pathway Studio는 7.1 버전까지 업데이트 되었으며 꾸준한 기능 향상과 데이터베이스
업데이트를 통해 항상 최신의 데이터를 제공하고 있다. Pathway Studio는 다양한 연구 분야를 비롯하여 제약회사를 포함한
전세계의 고객에 의해 사용되고 있다. Ariadne사에서는 고객들이 Pathway Studio를 효과적으로 사용할 수 있도록
case study를 다양한 형태로 제공하기도 하며 국내에도 매 년 몇차례 방문하여 세미나를 개최한 바 있다. 앞으로도 지속적인
기능 향상과 최신의 데이터베이스를 유지함으로써 국내의 많은 연구자들에게 도움이 될 것으로 생각된다.
Posted by 人Co