- [학술자료] CLC Genomics Workbench 활용 사례 공유
- [학술자료] CLC Genomics Workbench 활용 사례 링크드인 공유 페이스북 공유 트위터 공유
-
[학술자료] CLC Genomics Workbench 활용 사례
엽록체 유전체 분석을 통한 두 약용 식물의 식별
참고 논문 정보
- 제목: Development of a Chloroplast-based InDel Marker that Discriminates between Paeonia suffruticosa and P. lactiflora
- 저자: Mi Sun Lee, Yi Lee (교신 저자)
- 출판지, 연도: Korean Journal of Medicinal Crop Science, 2022
- DOI: http://dx.doi.org/10.7783/KJMCS.2022.30.6.430
참고 논문
연구 개요
현대 생물학 연구에서 정확한 종 식별은 약용 식품의 품질 관리 및 보존에 필수적입니다. 충북대학교에서 진행된 연구에서 CLC Genomics Workbench를 활용하여 서로 매우 유사한 두 약용 식물, 모란과 작약을 구별할 수 있는 분자 마커를 개발하였습니다.
연구 방법 및 결과
연구팀은 CLC Genomics Workbench를 사용하여 두 식물의 엽록체 전장유전체를 비교 분석하였습니다. 이를 통해 in silico 상에서 P. suffruticosa와 P. lactiflora 사이의 여러 다형성 지역을 식별하고, 특히 InDel 마커인 PsPl-InDel-12를 개발하여 성공적으로 두 종을 구별할 수 있었습니다. 이 마커는 한국 전역에서 수집된 다양한 샘플에 적용되었으며, 각 종에서의 특이적 증폭 산물을 통해 정확한 종 구분이 가능하였습니다.
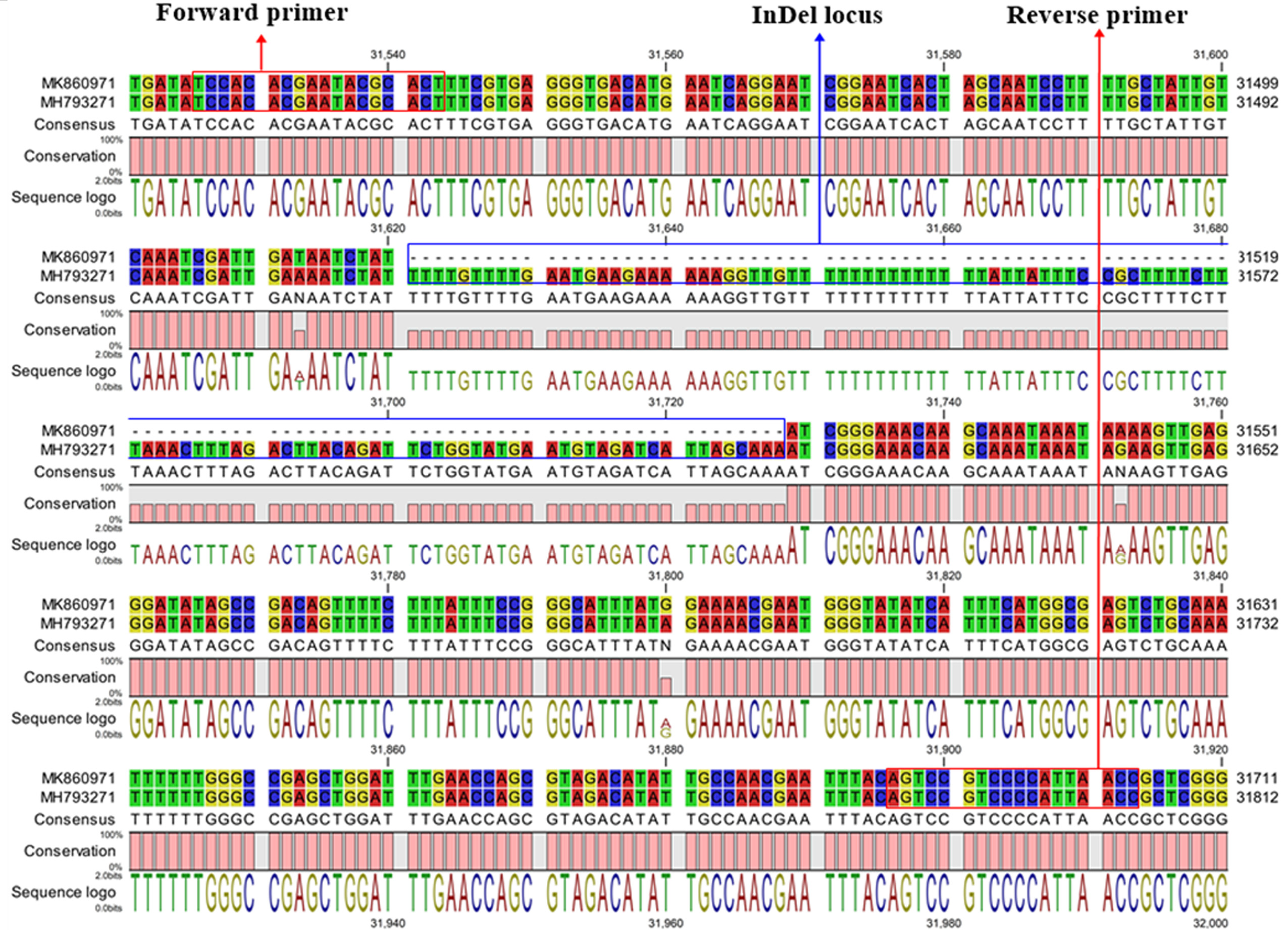
[그림 1] InDel locus sequence of the PsPl-InDel-12. Comparison of the chloroplast genomic information of P. suffruticosa (MH793271) and P. lactiflora (MK860971) with CLC Genomics Workbench.
연구의 응용
이 연구 결과는 약용 식물의 정확한 식별 뿐만 아니라, 향후 유사한 형태의 식물을 구별하는 데 CLC Genomics Workbench가 얼마나 효과적인지를 보여주는 예입니다. 연구자들은 이 도구를 사용하여 생물 다양성 연구, 자원 관리, 그리고 약용 식물의 품질 보증에 필수적인 정확한 데이터를 확보할 수 있습니다.