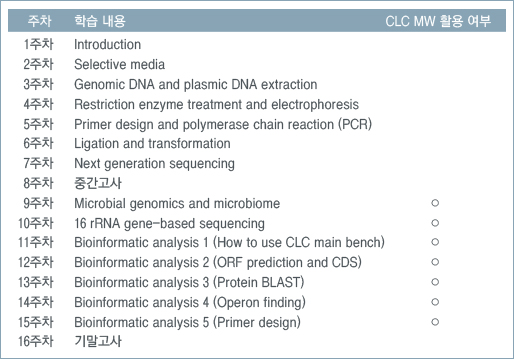
[LAB.STORY 02] 충북대학교 특용식물학과 생명공학 연구실 / 건국대학교 의생명공학과 미생물대사공학 연구실
- Posted at 2019/02/15 14:21
- Filed under LAB.STORY
Office 솔루션의 특징은 진입장벽이 낮고 문서 및 data를 구성원 간에 공유하고 자유롭게 수정할 수 있으며, 해당 기능으로 인해 업무 효율성에 시너지를 생성시킵니다.
그러나 안타깝게도 생물학 연구실에서 생성되는 일부 data들은 office 솔루션으로 저장하고 가공하기에는 어려운 것들이 있습니다.
예를 들어 염기서열에 경우 기본적으로 A, T, G, C로 구성되어 있어 문서파일로 저장할 수는 있지만 유의미한 정보를 알아보기 어려우며 가공하기 또한 어려운 것이 현실입니다.
이는 생물정보 data 전용 솔루션이 있으면 쉽게 해결할 수 있습니다. CLC Main Workbench는 Bioinformatics 분야에서 염기서열 분석을 위한 가장 기본적인 소프트웨어로 DNA, RNA, Protein, Digital Gene Expression 등의 분자생물학 데이터를 통합 분석 할 수 있습니다.
이를 통해서 연구실 단위에서 생물정보 분석 및 공유를 원활하게 진행할 수 있는데요, 실제 연구실에서 개인 PC 단위로 설치하여 활용하고 있는 사례를 소개해 드릴까 합니다.
충북대학교 특용식물학과 생명공학 연구실 (이 이 교수님)

이이 교수님의 지도하에 인삼, 당귀, 더덕, 도라지, 잔대, 대추나무, 소나무, 은행나무 등 다양한 약용식물 및 특용식물의 유전정보를 바탕으로 우수품종 육성 및 식물의 종 판별, 유전 다양성 분석 등을 위한 분자표지 개발 및 약용식물에서 주요 약리성분 중 하나인 사포닌의 생합성에 관련하는 유전자 탐색 및 역할 구명 연구를 수행하며 다양한 작물에 대해 RNAi를 이용한 형질전환 식물 개발연구 등을 수행하는 연구실입니다.
저희 실험실에서는 오래전부터 CLC Genomics Workbench와 CLC Main Workbench를 사용해오고 있습니다. CLC Main Workbench는 바이오인포메틱스 전문가가 없는 일반 연구실에서 쉽게 DNA 염기서열과 아미노산 서열 등을 이용하여 클로닝, PCR, 계통분석 등을 할 수 있고, CLC Genomics Workbench의 경우 다양한 플랫폼으로 생산된 NGS 데이터를 활용하여 trim, assembly, Blast 등을 수행할 수 있었습니다.
이 프로그램의 가장 큰 특징은 Windows 환경에서 'GUI를 통해서 구동되므로 다양한 명령어를 숙지하고 있을 필요가 없어서 누구나 쉽게 배울 수가 있고, 속도가 매우 빠르고 적은 양의 메모리를 사용하여 그다지 용량이 크지 않은 메모리를 가지고 있거나 그리 빠르지 않은 CPU를 가지고 있는 PC에서도 구동이 가능하다는 점'입니다. 특히 우리 연구실처럼 '여러 개의 개체로부터 NGS데이터를 얻어서 비교분석을 하는 경우에 개개의 분석을 매번 하지 않고 batch mode를 사용하여 여러 개의 데이터를 순차적으로 분석하여 연구자의 시간과 노력을 아낄 수 있게 해주는 편리한 프로그램이었습니다.
따라서 이 프로그램을 활용한다면 포스트게놈 시대에 다양한 형태의 게놈 데이터를 이용할 수 있어서 연구실의 연구 능력을 한층 업그레이드시킬 수 있다고 생각됩니다.

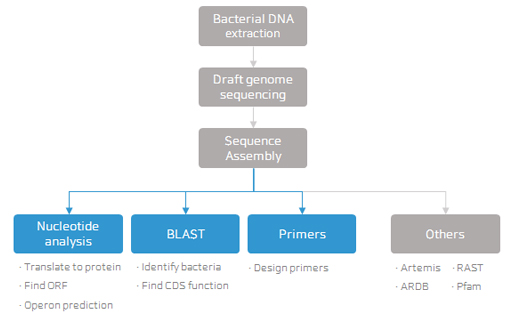
지노믹스 방법을 이용하여 미생물로부터 신규화합물을 찾아내고 이를 의약품으로 응용하는 연구 및 임상에서 쓰이고 있는 값비싼 단백질기반 의약품을 미생물에서 대량으로 생산할 수 있는 플랫폼을 개발하는 연구실입니다.

DNA 염기서열 기술이 발달하면서, 증가하는 생물정보의 데이터를 효율적으로 분석 및 관리하는 시스템의 필요하였습니다. 특히, 연구를 진행함에 있어 방대한 데이터베이스 내에서의 자료수집은 연구 결과와 연결되는 중요한 부분이라고 할 수 있습니다. CLC bio사에서 제공하는 CLC Main Workbench의 경우, 웹 기반 데이터베이스 내 검색, DNA 염기서열 분석 그리고 분자생물학 클로닝(cloning) 등의 다양한 분석 기능을 제공하고 있습니다. 그뿐만 아니라, '염기서열을 비교 분석한 결과를 이미지화하여 데이터 저장이 가능해 다른 연구자들과 정보를 공유할 수 있어 데이터를 손쉽게 다룰 수 있다는 점이 강점'이라고 생각합니다.
분자생물학을 기반으로 실험을 수행하는 연구실에 있어 'DNA 염기서열의 유전자 정보를 annotation 하는 것은 정말 중요한 일이라고 할 수 있습니다. 특히 염기서열 정보를 빠르게 수정 가능하며, Primer design과 molecular cloning tool은 실험을 디자인하는 데 있어 많은 도움이 되었습니다. 또, BLAST와의 연동 및 Sequence Alignment ' 를 활용한 계통수 분석의 사용은 실험 결과를 효율적으로 분석하는데 용이하였습니다. 앞으로도 CLC Main Workbench의 사용은 DNA 염기서열을 분석하고 이해하는 데 사용할 예정입니다.
이번 CLC Main Workbench의 사용은 생물정보 처리 시스템이 굉장히 중요하다는 생각이 들었으며, 효율적인 소프트웨어의 필요성을 느끼게 되었습니다. 생물학을 연구하는 과학자분들에게 CLC Main Workbench의 사용을 추천하려 합니다.
마치며
위의 두 연구실 모두 CLC Main Workbench를 통하여 연구실 내 생물정보 data 처리를 쉽게 저장하고 가공하여 연구를 원활하게 진행 중입니다.
생물정보 data 처리에 어려움을 느끼시거나 연구실 내에 data 포맷을 통일하고 싶으시다면 생물정보 전용 솔루션인 CLC Main Workbench를 한번 사용해 보시는 걸 추천해 드립니다.
마지막으로 사용 후기를 작성하여 주신 충북대학교 이이 교수님, 건국대학교 강학수 교수님께 감사의 말씀을 전합니다.
Posted by 人Co
- Response
- No Trackback , No Comment
- RSS :
- https://post-blog.insilicogen.com/blog/rss/response/304