Omics Technologies와 효과적인 in silico 테스트가 질병 치료를 위한 분자적 타켓 발굴을 가깝게 하고 있다?
- Posted at 2013/12/11 16:45
- Filed under 생물정보
연구팀은 파킨슨병에 관한 네트워크 뷰가 대표적인 신경퇴행성 질환인 치매 (Alzheimer)나 헌팅턴 무도병 (Huntingtun)과 같은 질병등에 광범위한 유전적 네트워크를 형성하는 기반이 될 수 있을 것이라고 발표하였습니다. 또한 분석을 통해 선별된 유전자들이 대표적인 신경퇴행성 질환의 공통적인 메카니즘과 관련이 있음을 확인하였습니다. 따라서 향후 이러한 네트워크 분석을 기반으로 하여 연구가 진행된다면, 파킨슨병과 같은 복잡한 신경 퇴행성 질환을 치료하기 위한 새로운 타겟이 될 수 있을 것이라고 발표하였습니다. 다만, 이러한 네트워크 뷰가 한정적이며, 부분적이어서 신경 퇴행성 질환과 같은 매우 복잡한 생물학적 pathway를 설명하기에는 부족하고, 또한 빙산의 일각에 불가하다고도 덧붙였습니다.
실제로 복잡한 생물학적 경로를 가지고 있는 다양한 질병들의 원인과 치료방법에 관한 수많은 연구가 진행되고 있는 현 시점에서 기존의 공개 정보들을 모아 잘 구성된 분석 프레임(well-made in-silico framework)에 적용한다면, 질병 치료를 위한 자체적인 파이프라인을 구축할 수 있지 않을까 하는 기대로 짤막하게 리뷰 하고자 합니다.
우선 논문에서는 파킨슨병 네트워크 뷰를 형성하기 위해 아래와 같이 워크플로우를 제시하고 있습니다 (그림 1).
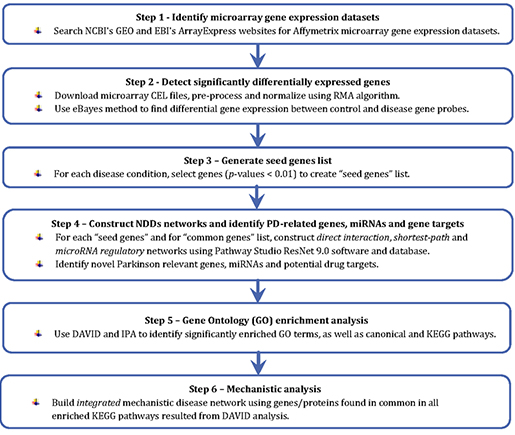
그림1.Study Workflow
Step1. 유전자 발현 데이터 셋 확인 (Identify microarray gene expression datasets): NCBI GEO 데이터 셋에서 diseased/control 상태의 원하는 샘플의 유전자 발현 정보 수집 [2].
Step2. 주요 DEGs 선별(Detection of significantly deferentially expressed genes): 데이터의 일관성을 위해서 동일한 방법으로 pre-preprocessing, normalizing, post-normalizing을 한 데이터를 선별하고, GEO데이터 셋에서 CEL 파일의 raw 데이터를 다운받아 R 프로그램으로 분석. 단, 선별된 모든 데이터들은 RMA(Robust Multi-array Average)[3], eBay(empirical Bayes)[39]통계 처리하여 주요 DEGs를 선별. (p-value<0.05)
Step3. "Seed genes" 생성: 연구팀은 GEO에서 3개의 데이터 셋 (GSE8397 HG-U133A, GSE8397 HG-U133B, GSE20295 HG-U133A)를 선별하여 중복되는 SDEGs들을 제거하고 각 조직별, 그리고, 조직에 상관없이 diseased/control 대비 DEGs 간의 공통 SDEGs 선별. (267개 선별, p<0.01)
Step4 . 파킨슨병과 관련된 새로운 유전자와 타겟 약물을 확인하기 위해 다양한 신경 퇴화질환 네트워크를 구축: PathwayStudio9.0 ResNet 9.0(2011.10.15 released) DB를 이용하여 신경퇴화 cell process와 관련된 direct interaction(DI), shortest-path(SP), common targets, 다양한 regulator를 분석하고, 분석된 정보로부터 신경퇴화 관련 유전자들과 선별된 267개 SDEGs 상관관계를 분석.
Step5. GO 분석: DAVID, IPA를 이용해서 GO 분석: Pathway Studio의 Pathway enrichment analysis를 통해 파킨슨병 pathway 분석
Step6. 기계론적 분석: DAVID 분석에서 얻어진 결과를 KEGG와 GO카테고리로 분석해서 공통으로 관련하고 있는 최종 46개의 유전자를 이용하여 질병 네트워크를 통합적으로 분석 [4].
선별된 유전자들 검증하기 위해서 워크 플로우를 통해 선별된 유전자들(267개)과 OMIM DB에 알려진 기존의 파킨슨병 주요 유전자 리스트를 비교한 결과, 누락된 몇몇 유전자들를 제외하고, 선별 유전자들이 기존 파킨슨병 주요 유전자들을 커버하고 있음을 확인하였습니다. 그리고, Pathway Studio외 DAVID, IPA 분석 등으로 최종적으로 46개의 유전자를 선택해서 파킨슨병 네트워크 뷰를 그렸습니다 (그림2).
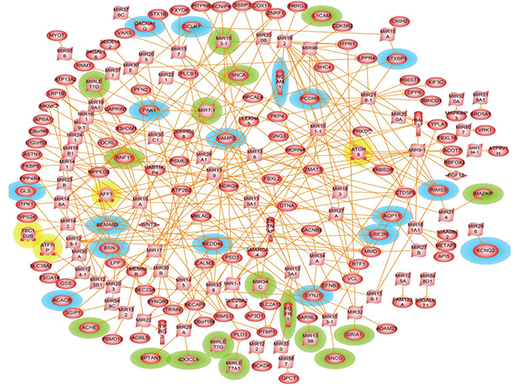
그림 2. Parkinson's disease regulatory network (blue(17): the genes of potential interest, green(7): miRNA)
특별히 더 주목할 만한 사항은 이러한 분석을 통해 새롭게 17개의 유전자들이 파킨슨병과 관련된 주요 유전자임을 확인할 수 있었다는 것과 이 17개의 유전자들이 질병 메카니즘 조절에 주요 역할을 하는 가장 근접한 pathway에서 발견되었다는 것입니다. 더불어, 기존에 밝혀진 miRNA[5]외에 추가적으로 7개의 microRNA를 더 확인할 수 있었고, 새롭게 밝혀진 miRNA와 mRNA와의 상관관계도 네트워크를 통해 확인할 수 있었다는 것입니다. 그리고, 이렇게 확인된 17개의 유전자들이 파킨슨병 뇌조직 뿐만 아니라, 치매 (Alzheimer), 헌팅턴 무도병 (Huntingtun)의 signal pathway에도 관여하여 모든 신경 퇴행성 질환에 공통적인 메카니즘을 설명하는 근거를 제시하고 있다는 것입니다. 그리고, 결론적으로는 연구팀이 발표한 파킨슨병 네트워크 분석을 통해서 파킨슨병을 촉발하는 분자 메카니즘(신경보호, 질병유발 등)에 관여하는 3개의 유전자(CX3CL1, SEMA6D, ILI2B)를 찾았으며, 최종 선별된 3개의 유전자가 관여하고 있는 신경보호, 질병 유발과 같은 분자 메카니즘의 향후 연구가 새로운 질병 치료를 위한 타겟이 될 수 있다고 제안하였습니다.
물론 이러한 in silico 상의 결론들은 많은 검증 절차가 필요하기 때문에 우리가 추구하는 최상의 결과를 제공하진 않지만, 이러한 분석적 시도는 다른 질병치료를 위한 연구에 적용할 만한 충분한 가치를 가진 듯 하였습니다. 그리고, 블로그 제목처럼 오믹스 정보들과 in silico 테스트가 질병 치료를 위한 해답에 점점 가까워지게 하는 것 같아 보이기 합니다. 빅데이터 시대에 수많은 불치병과 난치병이 이렇게 치료 방법을 찾아 가고 있지 않을까하고.....
모두가 그렇진 않지만, 저를 비롯한 많은 생물학자들은 각자가 가지고 있는 나무들을 모아 숲이 우거지길 기대하며 연구에 주력하는 듯합니다. 그러나, 포스팅한 논문과 같이 먼저 숲이 얼마나 우거져 있는지를 확인하고, 분류한 후, 가장 실한 나무를 찾는 연습과 훈련을 반복해서 더 효율적인 연구 결과를 얻는데 더 많이 주력해야 할 것 같습니다. 그리고, 그 중심에 생물정보가 있는 게 아닌가 하고 이 글을 쓰며 새삼 다시 생각하게 됩니다.
참고문헌
[1] Sreedevi Chandrasekaran a, Danail Bonchev, (2013) A Network View on Parkinson’s Disease, Computational and structural biotechnology J. 7(8)[2] Groettrup B. Boeckmann M, Strephan C., Marcus K., Grinberg LT, Meyer HE, Park YM (2012) Translational protemics in neurodegenerative diseases-16th HUPO BPP workshop Sep. 5, 2011, Geneva, Switzerland. Proteomics 12: 356-358
[3] Irizarry RA, Hobbs B, Collin F, Beazer-Barclay YD, Antonellis KJ, et al (2003) Explorarion, normalization, and summaries of high density oligonucleotide array probe level data. Biostatistics (Oxford England) 4: 249-264
[4] Kanehisa M. Goto S(2000) KEGG:kyoto encyclopedia of genes and genomes. Nucleic acids research 28:27-30
[5] Mouradian MM (2012) MircoRNAs in Parkinson's disease. Neurobiology of disease 46: 279-284
신가희 팀장
Posted by 人Co
- Response
- No Trackback , No Comment
- RSS :
- https://post-blog.insilicogen.com/blog/rss/response/143
Trackback URL : 이 글에는 트랙백을 보낼 수 없습니다


