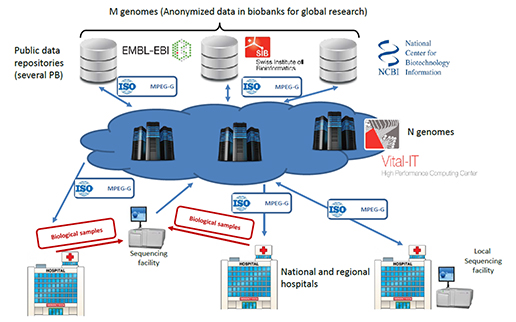
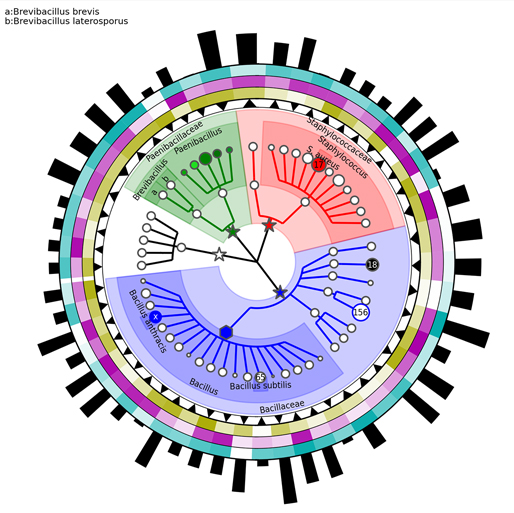
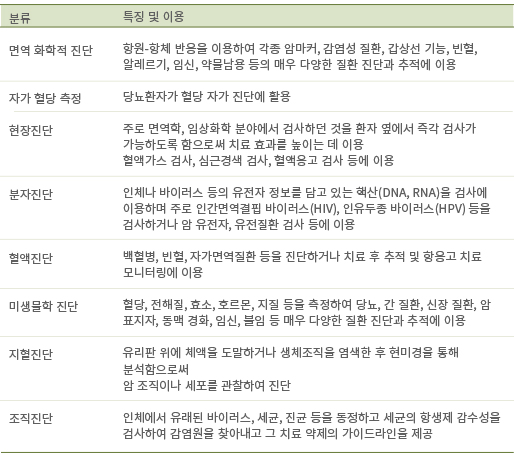
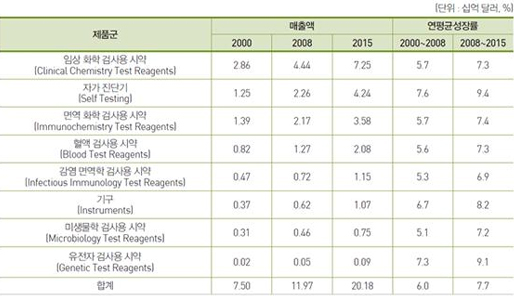
디지털 육종과 표현형 데이터 - 표현형 데이터가 디지털 육종 시대를 이끌다.
- Posted at 2021/04/11 14:38
- Filed under 생물정보

우리가 먹고 있는 청양고추, 사실은 독일 바이엘사에 로열티를 주고 종자를 구매 후 작물을 재배하여 섭취하고 있는 현실 알고 계셨나요? 그뿐만 아니라 제주 감귤, 파프리카, 고구마와 양파의 종자도 대부분 해외에서 사들여 먹고 있는 것입니다.
최근 세계적인 유전자 가위 업체들은 항산화·항노화 성분이 기존 콩보다 2배 이상 많은 콩을 개발해 미국 농무부(USDA)로부터 Non-GMO 판정을 받아냈다고 하는데요, 즉, 유전자변형식품이 아니라는 뜻으로 볼 수 있습니다.
이는 이미 선진화된 기술력을 가진 해외 기업들이 우리가 먹고 있는 식량을 좌지우지할 수 있는 여지가 더욱 커졌다는 말이기도 합니다.
다행히 우리나라에서도 디지털 육종 전환 사업이란 것을 통해 육종 기간을 6년에서 3년으로 줄이고 육성 품종의 상품화율을 5%에서 50%로 대폭 끌어올리는 것을 목표로 국내 종자 산업 첨단화를 위한 사업이 진행 중인데요,
이 디지털 육종에 표준화되고 객관화 된 표현형 데이터가 매우 중요합니다.
디지털 육종과 표현형 데이터? 다소 생소하게 느껴질 수 있겠지만, 블로그를 통해 그 궁금증 하나둘씩 해결해드리고자 합니다.

기후 및 환경 변화 등의 이유로 우수한 신품종을 빠르게 육성하기 위해서는 종묘단계에서 우수한 개체를 선발하는 것이 무엇보다 중요한데 이를 위해 경험 중심의 전통육종[1]에서 빅 데이터 기술과 인공지능기술(AI)로 신속한 의사결정이 이뤄지는 디지털육종[人Co블로그:https://www.insilicogen.com/blog/370]으로 전환이 필수적으로 요구되고 있습니다.
최근 유전자분석 기술(NGS)의 도움으로 유전체 정보는 폭발적으로 증가했으나 표현형[2] 정보의 측정 및 분석기술에 한계가 있다는 점이 육종 기술 도입의 한계로 지적되었습니다.
그러나 최근 RGB, NIR 카메라 및 영상 기술이 발달함에 따라 이를 이용해 크기, 수, 이상 현상 등 작물의 표현형을 정확하고 빠르게 대량으로 수집할 수 있게 되었고, 수집된 데이터를 바탕으로 다양한 기술을 이용해 영상정보(사진, 양상)를 디지털화하고 우수 경제 형질과 연계해 우수한 품종을 선발하는 표현체 이용 기술이 주목을 받고 있습니다.
이러한 표현체 연구는 작물의 형태적 특징을 영상 기술을 통해 수치화 및 객관화하여 분석하는 기술로써 표현형과 연관된 유전자의 연관 관계를 밝혀 그 특성을 이용한 우수 품종 개발을 지원할 수 있는 아주 유용한 방법이라 할 수 있습니다. 즉, 각 개체의 표현형질과 유전적 특성을 미리 알 수 있어 육종 시 원하는 형질을 가진 모본과 부본 간의 교배가 가능하므로 우수 경제 형질을 가진 개체를 선발할 수 있으며 육종 기간 단축을 통해 노력과 시간의 감소 효과를 얻을 수 있습니다.
따라서 전통농업기술과 분자육종, ICT 및 인공지능 등 첨단 기술을 접목한 표현체 연구를 기반으로 차세대 디지털육종 시스템으로 변환을 통해 육종 기간 단축과 우수후보 발굴 등 신품종 개발 효율성을 높일 수 있습니다.
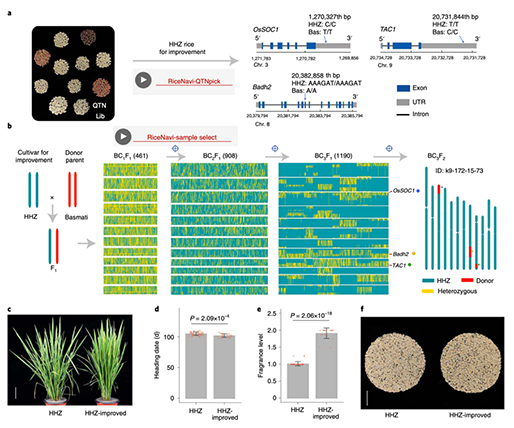

- 데이터베이스 구축 단계
- 각 재배 및 사육 단계에서 육안 또는 영상, ICT 장비를 이용해 데이터를 체계적으로 수집하는 단계로, 데이터를 수치화하고 객관화시켜 신뢰할 수 있는 표현체 빅데이터를 구축해야 함.
- 기온, 습도, 날씨 등 다양한 환경 요소를 비롯하여 작물의 성장과 발달 단계에 맞는 정확한 정보(온톨로지)를 통해 수치화하여 수집하는 단계.
- 지식정보 그래프 구축 단계
- 수집 데이터를 '노드'-'엣지'의 그래프로 구현하는 단계로 서로의 데이터 관계를 명확하게 정의하여 표현형, 기능, 유전형의 관계를 체계적으로 구축하는 단계.
- 연관 관계 분석 단계
- 각 수집 요소에 대하여 알고리즘을 적용하는 단계로 표현형 및 환경정보와 유전형의 연관 관계에 대하여 중요도를 부여하여 특정 표현형에 대한 쿼리 결과의 순위를 제공하는 단계로 이를 뒷받침하는 유전형의 정보도 제공.
- 맞춤형 분석 단계
- 데이터베이스를 재구성하거나 탐색을 통해 자신과 가진 데이터를 비교하거나 분석할 수 있는 플랫폼을 제공하는 단계로 특정 표현형에 대한 집단 비교 분석 및 AI 기술 도입을 통해 최적의 교배 지침을 제공하는 단계.
기존 전통육종에서 디지털육종으로의 전환을 유도할 수 있는 표현형 데이터는 유전체 데이터와의 결합과 AI 기술 적용을 통해 새로운 미래 먹거리를 발굴에 활용할 수 있습니다.

그러나 이러한 표현형 데이터는 무엇보다 표준화와 객관화가 중요합니다. 특히 단위는 무척이나 중한데 이를 간과한 사례가 있습니다. 단위가 헷갈려 1,400억 원짜리 우주선이 폭발한 사례[중앙일보]에서 보는 것처럼 주요 단위인 야드와 미터법의 혼동으로 이와 같은 엄청난 피해를 남겼습니다. 표현형을 수집할 때 정확한 용어와 단위 사용은 필수 요소입니다. 이렇게 체계적으로 수집된 표현형 데이터가 유전형 데이터를 만났을 때 진정한 위력을 발휘할 수 있는 것입니다.



(출처:다양한 커피잔[Cyril Saulnier 제공)

이를 위해 공공기관과 민간 기업에서 홍수처럼 쏟아지는 많은 양의 데이터를 적절히 관리하고 분석할 수 있도록 빅데이터 구축 사업을 꾸준히 진행하고 있는데요, 디지털 육종을 위한 표현형 데이터도 데이터베이스 구축을 통해 그 활용도와 효용성을 극대화할 수 있을 것입니다.

- 표현형
표현형은 어떤 생명체의 겉으로 관측이 가능한 특정 모습이나 성질을 의미하며 유전형과 반대되는 개념.
멘델의 완두콩 실험을 설명하기 위해 처음 표현되었으며, 현대에 이르러서는 그 개념이 크게 확대되었습니다. 초기 멘델이 형질의 특성을 설명하기 위해 사용된 이 개념은 '유전자형이 곧 표현형으로 드러난다' 는 개념이었으며 완두콩의 '동그랗다'와 '주름지다' 등과 같이 실제 겉으로 드러나는 모양을 표현형이라고 부를 수 있습니다.
표현형은 우리가 흔히 마주할 수 있는 머리카락 색, 눈 색, 키 등과 같은 외향적인 모습뿐만 아니라 특징적인 행동, 발생, 생리학적 특성 또한 포함합니다. 이러한 표현형은 초기 유전형에 의해 결정되며 향후 환경적 요인에 의해 변할 수도 있습니다.
- 육종의 개념
육종이란 농작물이나 가축을 개량하여 경제(실용) 가치가 더 높은 새로운 품종을 개발하고 증식하여 보급하는 기술입니다. 육종의 목표는 수량 증대와 품질 향상, 내재해성, 내병성, 맛, 향기(풍미), 모양, 사육 환경 등이 다양한 경제 형질로 정해질 수 있습니다.
육종의 대상은 농경을 시작한 이래로 산업적으로 유용한 형질(표현형)을 가진 모든 생물체가 그 대상이었습니다. 경주 능력을 목표한 '서러브레드' 경주마, 우리가 즐겨 먹는 마블링이 우수한 1등급 '한우', 매운맛의 강자 '청양고추', 가난에서 벗어나게 해준 수확량의 제왕 '통일벼', 밀을 대체할 벼 품종 '가루미' 등 동식물을 망라하고 인간에게 유용한 경제 형질을 가진 모든 분야에서 육종이 이루어져 왔습니다.
이렇게 다양한 특징을 가진 농작물 또는 가축을 만드는 것이 전통적인 분리육종만으로는 수십 년 이상을 필요로 하므로 현대 육종방법에서는 최첨단 과학기술을 사용하여 종자 개발을 진행하고 있습니다.
- 전통육종과 분자육종
전통 육종 - 직접 식물 또는 동물을 교배하고, 재배(사육)하여 선발한 개체를 다시 재배하여 확인 작업을 거쳐 품종화시키는 방법이기에 최소 7년에서 20년 이상의 시간이 소요되며 겉으로 드러난 표현형만을 기준으로 하므로 육종의 목표가 되는 형질 외에 다른 형질의 내재성을 모르는 등 그 한계가 분명합니다.
분자 육종 - 육종기술에 분자 마커를 활용한 분자생물학 기술을 접목한 새로운 육종방법으로 마커를 통해 각 개체의 유전적 특성을 규명하고 효율적으로 우수한 개체를 판별하는 기술입니다. 유용한 형질을 가진 개체를 찾고 교배하는 것은 전통과 같으나 자손 세대의 재배 없이 분자마커를 이용해 원하는 개체를 찾을 수 있으며 많은 시간을 단축하거나 눈으로 확인할 수 없는 경우 등 개체 선발에 도움을 줍니다.
Posted by 人Co
- Tag
- 교배육종, 데이터육종, 디지털육종, 분자육종, 아이브리딩, 육종, 전통육종, 표현형데이터
- Response
- No Trackback , No Comment
- RSS :
- https://post-blog.insilicogen.com/blog/rss/response/377