기생충과 우리와의 관계를 유전체적으로 살펴본다면....
- Posted at 2013/11/20 14:10
- Filed under 생물정보
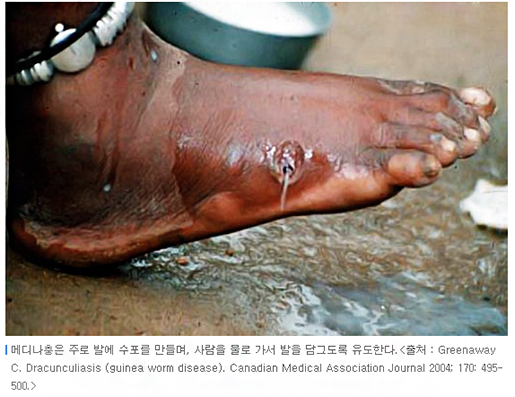
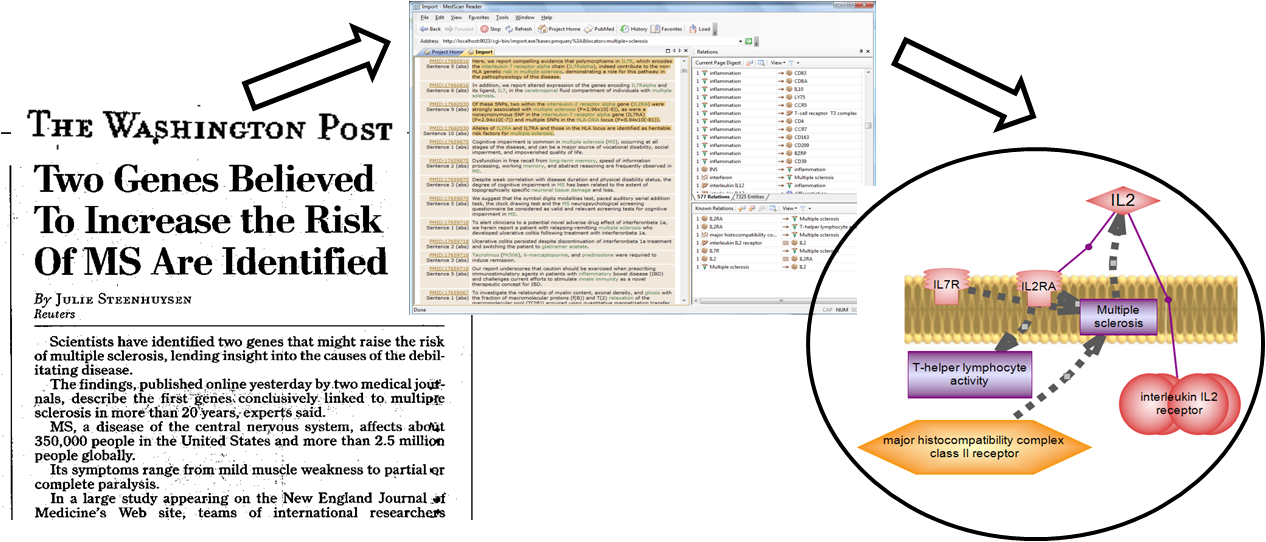
일례로 의학을 상징하는 카두케우스의 지팡이에 감긴 뱀 두 마리는 메디나충을 의미하는 것으로 예로 부터 알려진 대표적인 숙주를 조종하는 기생충입니다. 아프리카등지에서 오염된 물을 통해 감염된 메디나충은 성충이 되어 숙주 몸 밖으로 안전하게 알을 배출하기 위해 숙주인 사람을 물가로 이끌게 합니다. 중력 때문인지, 물과 근접하기 위함인지 정확하진 않지만, 대부분 물과 쉽게 닳는 발이나 복숭아뼈 근처 피부를 뚫어 수포를 만들고 염증반응을 유발하여 화끈거리는 발을 물가로 인도 하는 메디나 충은 그 긴 몸을 드러내고 본인의 목적을 달성합니다. 다시금 중간 숙주인 물벼룩의 먹이가 되어 보다 많은 종 숙주(사람)에서 삶을 이어갈 수 있도록 말입니다.
기생충은 오로지 자손 번식을 위해서만 살아간다고 합니다. 그렇게 진화된 것이라고 합니다. 회충은 몸의 대부분이 생식기로 구성되어 있으며 암컷 회충은 하루에 20만개의 알을 낳는다는 것도 이런 이유일 것입니다. 유충 상태로 우리 몸 어딘가에 정착된 기생충은 처음 직면하는 어려움이 우리 몸의 면역 세포들과의 소소한 싸움입니다. 면역 세포에 비해 덩치가 워낙 큰 기생충은 사실상 상대가 되지 않을 만큼 면역세포들에게는 큰 적입니다. 그러나 소소한 면역 세포의 끊임없는 괴롭힘은 사실 기생충으로 하여금 '나는 오직 자손 번식만 하면 돼..너 괴롭힐 맘 없으니 잠시 방 하나만 내어주면 조용히 지낼께~' 라며 양해를 구하게 하고, 싸우기 힘든 면역 세포 입장에서는 '그래, 약속만 한다면야...내가 참아주지.' 라며 서로 합의에 이르게 합니다. 그러나 이러한 합의는 성충이된 기생충의 살아가는 목적인 알을 안전하게 다음 숙주로 옮겨주어 자손을 널리널리 퍼뜨려 종족이 사라지지 않도록 하기 위해 위반하게 되고 숙주에게 해를 끼치게 됩니다. (물론, 중간 중간 이상한 물질을 분비하여 신경계를 교란 시키거나 여러 조직을 돌아다니면서 물리적으로 치명적인 해를 입히는 기생충도 많이 있습니다. )
여기서 생물을 연구하는 우리는 의문을 품었습니다. '기생충과 면역 세포간의 합의가 이뤄진 그 기간 동안 민감했던 면역 시스템이 대충 눈감아 줄 정도로 유연해 졌다면, 아토피나 알레르기와 같은 예민한 자가 면역이 호전 되지 않을까?' 전 세계 많은 과학자들은 실험을 했고, 결론은 '기생충이 아토피와 같은 자가 면역 질환에 도움이 된다'를 주장 하는 논문과 '유의한 결과가 없다'를 주장하는 논문이 함께 맞서고 있습니다[1][2][3][4].
이러한 상황을 생물정보학적으로 접근해 보겠습니다. 현재까지의 자가면역 질환에 대한 기생충의 효과는 대부분이 자가면역질환을 유도하는 면역 시스템인 Th2 면역반응에 수반되는 IL-4, IL-5, IL-6, IL-10, IL-13와 같은 cytokine과 IgE와 같은 염증반응을 유도하는 면역 글로빈의 반응을 중심으로 연구 되었습니다. 이를 유전체 전반으로 확장하여 생각해 보면, 실험의 대상이 되는 숙주인 사람은 생김새 부터 식습관, 성격까지 다양한 나름의 특징을 가지고 있고 이는 46 개 염색체 전반에 분포하고 있는 유전체상의 서열변이로 인해 갖는 다양성입니다. 따라서, 본래 태어날 때부터 갖는 외부 자극에 대한 면역 반응이 서로 다를 수 있으며 자라온 환경적인 이유로도(Epigenomics) 그 반응이 상이 할 수 있습니다. 같은 아토피 환자라 할지라도 반응 정도, 반응 물질, 반응 시기까지 모두 다양하게 나타나는 예가 바로 이에 해당된다 할수 있습니다. 따라서 복잡한 면역시스템과 기생충간의 상호 대응 관계를 일반화 시키기 위한 조건을 잡는 것은 쉬운일이 아닐것입니다.
생물정보분야에서는 이러한 복잡한 배경속에서 결론을 얻기 힘든 상황을 해결하기 위해 시맨틱 모델을 적용한 데이터 베이스를 활용하기
시작했습니다. 최대한 다양한 정보를 바탕으로 각각의 경우에 따른 반응정도를 정리하는 데이터 베이스를 구성하는 것으로 다음과 같은
정보들을 정리 할 수 있습니다.
첫번째, 유전체내 서열 변이 정보를 정규화 하여 데이터 베이스화 합니다.
예를 들어 집먼지 진드기에 알레르기를 일으키는 유형과 꽃가루 혹은 견과류에 반응을 보이는 유형 각각의 유전체 정보를 정규화
합니다. 단일염기변이(SNPs) 정보, gene loss 정보와 같은 유전체 전반에 걸친 정보를 데이터베이스화 합니다.
두번째, 이들의 유전자 발현 패턴을 정보화 합니다.
동일한 외부 자극에 대한 반응의 정도를 각각의 유형(집먼지진드기, 꽃가루, 견과류 반응)에 따라 유전자의 발현 패턴을 정규화
합니다. 또한 기생충(유충) 감염 상태에서의 유전자 발현 정도도 함께 체크합니다. (단, 감염 초기 4주 안에는 숙주에 큰 피해를
주지 않는다는 안정성 테스트가 있었습니다[5].) 뿐만 아니라 감염되는 기생충의 종, 감염 기간, 감염량에 따라서도 많은 차이를
보이므로 이들에 대한 적절한 실험 디자인이 필요합니다.
세번째, 기본적인 유전자의 생물학적 기능 정보를 비롯해 현재 알려진 세포내 신호전달 정보를 데이터베이스화 합니다.
KEGG를 비롯한 biocarta의 데이터베이스와 현재까지 업데이트 되지 못한 문헌상의 pathway 정보를 데이터베이스화 합니다(문헌상의 pathway 정보를 텍스트마이닝을 통해 정리해 주는 프로그램은 'PathwayStudio', 'Ingenuity' 등이 있습니다).
나이, 체중, 성별, 가족관계, 혈액형, 성격, 질병이력 등을 비롯해 식습관, 사는곳 등 가능한 많은 표현형 정보와 환경적인 요인을 정보화 합니다.
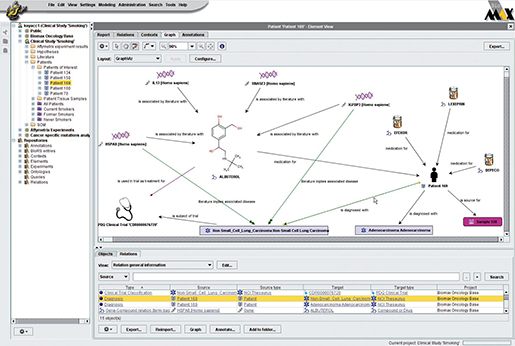
다섯번째, 현재까지 정리된 데이터들을 서로 연결합니다.
이때,
각각의 정보에 대한 의미론적인 관계(Semantic model)를 맺어주게 되고 이를 통해 기계가 특정 자극에 대한 결과를
통합적으로 살펴볼수 있도록 합니다(이를 위해서는 Biomax사의 'BioXM' 을 이용할수 있습니다).
이렇게 구축된 데이터베이스는 사용자의 질문에 대해 가능한 모든 정보를 배출하게 됩니다. 따라서 사용자는 최대한 많은 경우를 고려한 질문을 데이터베이스에 요구하여 데이터베이스로 하여금 시맨틱모델이 적용된 많은 조건을 모두 통과한 결과를 배출 하도록 하는 것이 필요합니다. 또한 의미있는 결과를 얻기 위해서는 앞서 언급했듯이 이러한 시맨틱 데이터 베이스가 최대한 많은 정보를 담아야 합니다. 따라서 다양한 실험 결과를 생산해 냄과 동시에 이미 공개된 데이터와 세계 각국에서 따로따로 진행된 일부 정보들을 모두 활용하여 세포속 네트워크 처럼 만들어 가는 것이 가장 중요하다 할 수 있습니다.
현재까지는 열거된 정보를 담은 기생충과 숙주와의 관계를 살펴 볼수 있는 데이터베이스를 찾기가 쉽지 않습니다. 그러나 시간이 갈수록 촘촘해지는 네트워크를 구축해 간다면,"땅콩에 예민한 알레르기 반응을 보이는 반면 집먼지 진드기에는 반응을 보이지 않고있는 10세 이하의 어린이들 중 Heligomosomoides polygyrus 감염후 IgE 항체의 양이 4배이상 증가하는 숙주들의 유전자 loss들이 관여하고 있는 공통된 pathway는 무엇인가?" 라는 질문에 대한 정보에서 재밌는 단서를 찾을 수 있지 않을까 기대해 봅니다.
Reference
- Fallon PG, Mangan NE (2007) Suppression of TH2-type allergic reactions by helminth infection. Nat Rev Immunol 7: 220-230.
- Yazdanbakhsh M, Kremsner PG, van Ree R (2002) Allergy, parasites, and the hygiene hypothesis. Science 296: 490-494.
- Harnett W, Harnett MM (2008) Therapeutic immunomodulators from nematode parasites. Expert Rev Mol Med 10: e18.
- Yazdanbakhsh M, van den Biggelaar A, Maizels RM (2001) Th2 responses without atopy: immunoregulation in chronic helminth infections and reduced allergic disease. Trends Immunol 22: 372-377.
- Falcone FH, Pritchard DI (2005) Parasite role reversal: worms on trial. Trends Parasitol 21: 157-160
작성자 : Codes 사업부 Research팀
신윤희 선임
Posted by 人Co
- Tag
- 기생충, 네트워크, 단일염기변이, 데이터베이스, 시맨틱, 시멘틱, 유전자발현, 유전체
- Response
- No Trackback , No Comment
- RSS :
- https://post-blog.insilicogen.com/blog/rss/response/142