NGS는 무엇이고, 어떻게 분석해야 할까요?
- Posted at 2012/03/29 17:22
- Filed under 제품소식
◆ Next Generation Sequencing?
DNA 염기서열의 정보는 그 동안 sanger에 의해 개발된 방법을 자동화하여 DNA 가닥에서 A, T, G, C의 순서를 빠르고 정확하게 읽어내는 캐필러리 장비(Sanger sequencing, 1세대 시퀀싱)를 이용하여 분석하였고 유전자의 발현, 다양성 및 상호작용 등의 정보로서 활용할 수 있어 굉장히 중요합니다.
따 라서 많은 염기서열을 저렴한 비용에 수행할 수 있는 기술의 필요성이 증가되면서 차세대 염기서열 분석 기술(Next Generation Sequencing, 2세대 시퀀싱)을 이용한 플랫폼들이 소개되어, 생명과학 분야에 있어서 특히 유전체학 분야에 큰 영향을 끼치고 있습니다.
또 한 현재 염기서열 분석 기술은 더 짧은 시간에 더 적은 비용으로 더 많은 염기서열을 결정할 수 있는 플랫폼 장비들이 계속적으로 탄생되어 시퀀싱 chemistry 차이에 따라 차세대(2세대), 3세대, 4세대로 분류하여 부르기도 하면서 비약적인 발전을 하고 있습니다.
◆ NGS 데이터 분석 도구
현재 생산되는 NGS 데이터는 장비가 점점 발달함에 따라 한 번 플랫폼을 run하여 얻는 데이터양만 해도 어마어마합니다. 따라서 이러한 데이터를 한꺼번에 분석하려니 그에 맞는 메모리 및 스토리지 등의 하드웨어 사양의 고려와 또한 생물정보를 알고 있지 않는 이상 명령어 방식의 커맨드라인의 툴을 이용하기란 쉽지 않습니다.
NGS 데이터의 분석 단계는 크게 pre-processing, assembly 그리고 assembly를 이용한 이차 분석으로 나누어집니다. Pre-processing 단계에서는 다양한 플랫폼으로부터 single reads, long reads, paired-end reads 등 시퀀싱된 reads의 정보를 assembly 단계에 적용하기 위한 작업을 수행하고, 분석의 방향과 목적에 맞는 assembler를 선택하여 assembly를 수행하게 됩니다. 이 후 assembly 결과를 이용한 variation 분석, expression 분석, binding site 분석 및 전체 정보에 대한 브라우저 구축 등 다양한 이차정보를 분석하게 됩니다.
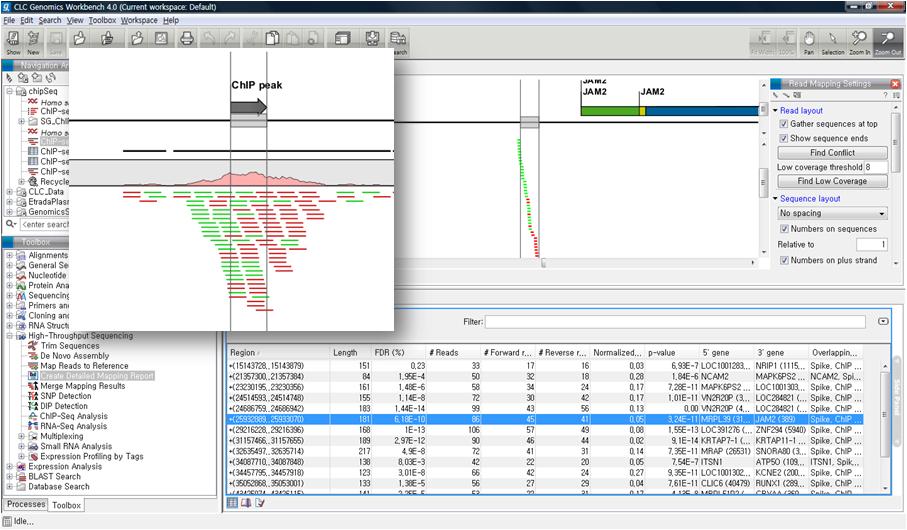
이 러한 분석 단계들을 하나의 툴에서 모두 진행하고 그 결과를 그래픽하게 확인할 수 있다면 NGS 데이터를 다루는 생물학자들이 무척이나 수월하게 연구를 수행할 수 있을 것입니다. 이러한 목적으로 개발된 NGS 데이터 분석 도구 중의 하나인 CLC Genomics Workbench를 소개하고자 합니다.
◆ CLC Genomics Workbench의 응용
CLC bio사의 CLC Genomics Workbench는 그래픽 인터페이스 기반의 NGS 데이터를 분석하기 위한 데스크탑 솔루션입니다. 현재 Roche 454, Illumina, Applied biosystems, Helicos, Ion torrent 등 다양한 회사의 NGS 플랫폼 장비에서 생성되는 모든 시퀀싱 데이터의 분석을 지원합니다. 또한 기존 sanger 데이터를 비롯하여 각 플랫폼에서 제공하는 다양한 데이터 셋을 hybrid하여 assembly를 수행하고 그 결과를 그래픽한 결과로 확인할 수 있습니다.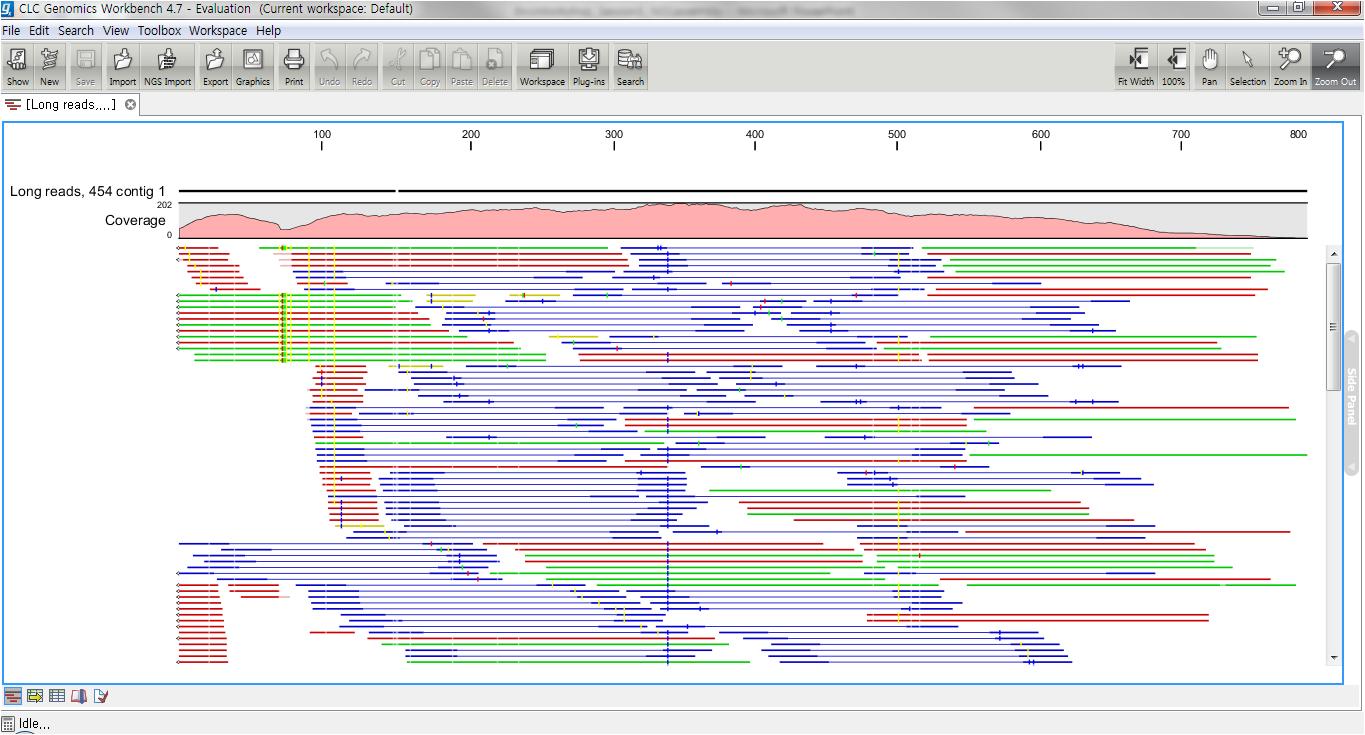
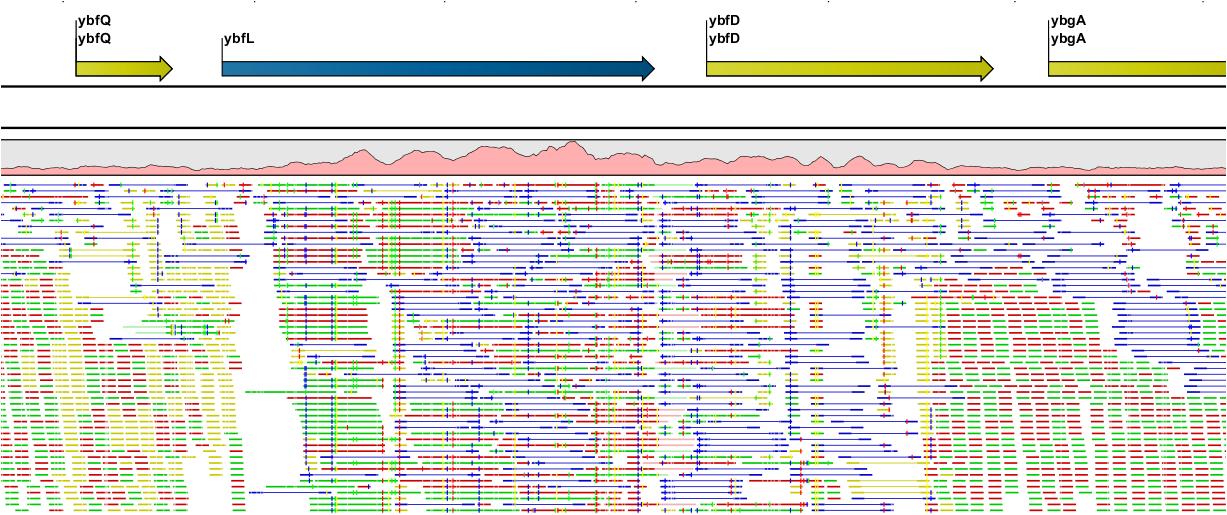
CLC Genomics Workbench에서 제공하는 assembly는 SIMD(Single Instruction Multiple Data) 기술을 적용하여 병렬연산으로 막대한 양의 NGS 데이터를 빠른 속도로 분석할 수 있어 유전체 크기에 관계없이 분석이 가능합니다. 단 많은 데이터를 분석할 시 고사양의 하드웨어 성능도 필요합니다.
이 렇게 NGS를 통하여 전체 염기서열 결정 및 re-sequencing을 통한 유전체 상의 여러 가지 변이 연구가 활발해졌으며 보통 NGS를 이용한 variation 연구는 유전체 시퀀싱을 진행하여 reference 서열에 정확한 맵핑과 정렬을 통하여 비교하고 있습니다. CLC Genomics Workbench는 일차적으로 assembly를 수행하고 이 후 서열 간의 비교 분석을 통한 SNP 및 small Indel 분석을 진행할 수 있습니다.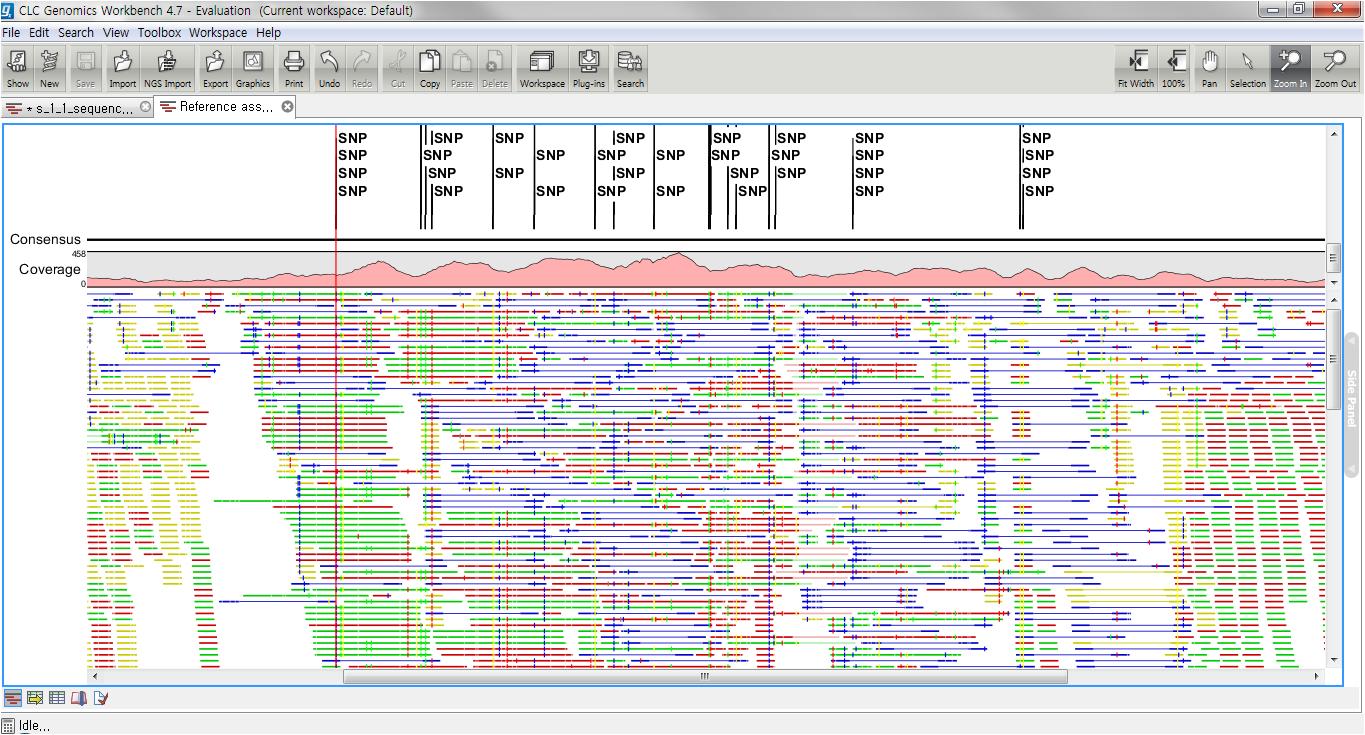
codes@insilicogen.com
많은 생물학 연구자 분들에게 NGS 분석 방법에 대한 이해를 도울 수 있으면 좋겠습니다.
Posted by 人Co
- Response
- No Trackback , No Comment
- RSS :
- https://post-blog.insilicogen.com/blog/rss/response/103
Trackback URL : 이 글에는 트랙백을 보낼 수 없습니다


